Oleh: David. L. Suarez
Diterjemahkan oleh: Agna D. Lantria
DISCLAIMER
This article was translated to enable Bahasa Indonesia readers to better understand the topic explained inside the article in their native language. The translator made no profit in translating the article.
Pengenalan
Virus Influenza A (IAVs) adalah patogen yang penting bagi dunia kedokteran hewan dan manusia. Kategori virus tersebut memiliki cakupan hospes (inang perantara) yang luas, termasuk di dalamnya sejumlah besar spesies unggas dan mamalia. Epidemiologi dan ekologi virus Influenza A sangat kompleks, melibatkan berbagai hospes unggas liar, unggas tangkapan yang dibesarkan di penangkaran, maupun unggas yang diternakkan, selain juga melibatkan berbagai spesies hewan mamalia liar maupun domestik di berbagai kondisi lingkungan yang berbeda seperti manusia, babi, kuda, anjing dan kelelawar. Selain itu juga terdapat beberapa infeksi sporadis pada berbagai hospes mamalia (Gambar 1).
Karakteristik kunci lain yang dimiliki oleh virus ini adalah variasi genetik dan antigenik yang terjadi melalui kombinasi tingkat mutasi yang tinggi dan genome bersegmen (segmented genome) yang memungkinkan perubahan yang cepat dan beradaptasi terhadap hospes yang baru. Dalam keadaan yang tepat, virus ini dapat beradaptasi di hospes yang baru sedemikian rupa dimana ia dapat memperbanyak diri dan bertransmisi secara efektif sampai menjadi endemik pada spesies hospes baru tersebut. Pada umumnya, proses adaptasi ini memproduksi galur virus yang memiliki beberapa tingkat kekhususan hospes sehingga membuatnya lebih sulit untuk menginfeksi spesies yang lain. Sebagai contoh, virus yang telah endemik pada kuda menjadi kurang mampu untuk menginfeksi spesies lain seperti babi atau manusia. Batasan spesies ini bisa saja kurang jelas pada spesies unggas, dimana virus yang telah beradaptasi pada tubuh ayam biasanya juga mampu menginfeksi spesies yang secara genetik dekat dengan ayam, tetapi beberapa kelas bangsa unggas, seperti itik atau merpati bisa saja resisten terhadap infeksi virus dari ayam tersebut.
Infeksi Virus Influenza A menyebabkan berbagai macam penyakit klinis yang umumnya berhubungan dengan patogenesis dari virus, seperti misalnya beberapa virus hanya menimbulkan infeksi pada mukosa saja dan beberapa dapat menimbulkan infeksi sistemik. Kontrol virus pada hewan telah dilakukan menggunakan berbagai cara, diantaranya vaksinasi, karantina dan bahkan pemusnahan (focal culling) hewan yang terinfeksi. Tujuan pemusnahan virus di dalam populasi hewan dapat tercapai pada situasi tertentu, tetapi membutuhkan biaya yang tinggi. Pada banyak negara, virus ini telah menjadi endemik sehingga upaya kontrol dilakukan utamanya untuk meringankan kerugian secara ekonomi. Reservoir virus ini berada pada unggas liar, oleh sebab itu tujuan akhir eradikasi virus secara total tidak memungkinkan. Ini menimbulkan ancaman berkelanjutan akan potensi timbulnya virus baru yang unik dari unggas liar.
Etiologi
Klasifikasi
Virus Influenza Tipe A tergolong dalam keluarga Orthomyxoviridae, virus RNA dengan sense-negatif bersegmen. Orthomyxoviridae dibagi ke dalam enam genera yang diterima Komite Internasional Taksonomi Virus (International Committee on Viral Taxonomy), yang termasuk di dalamnya antara lain Virus Influenza Type A, Tipe B, Tipe C, Isavirus, Thogotovirus dan Quaranfilvirus [1]. Dua virus RNA baru telah diajukan sebagai genera potensial baru, yaitu virus Tipe D yang berhubungan dengan penyakit pernafasan pada babi dan ruminansia dan virus yang berhubungan dengan siklus kejadian kematian pada unggas eider/ itik laut eropa (Somateria mollissima) di Amerika Utara yang dinamakan Wellfeet Bay Virus [4, 23]. Virus Influenza Tipe A adalah yang paling tersebar luas dan paling penting diantara anggota Orthomyxovirus, menginfeksi banyak spesies unggas dan mamalia. Tipe B dan C merupakan pathogen pada manusia yang sangat jarang menginfeksi spesies lainnya, meskipun infeksi pada babi dan anjing laut telah dilaporkan [100]. Grup Isavirus mencakup diantaranya virus yang menyebabkan penyakit anemia infeksius pada ikan [61]. Thogotovirus merupakan arbovirus yang disebarkan melalui gigitan caplak telah diisolasi dari manusia dan hewan ternak [71]. Dan yang terakhir, Quaranfilvirus adalah virus yang berhubungan dengan caplak yang telah ditemukan pada manusia dan unggas [117]. Buku ini seterusnya akan memfokuskan diri pada pembahasan mengenai virus Influenza Tipe A pada unggas dan mamalia, tetapi juga akan memberikan sedikit gambaran mengenai virus Influenza B yang terkandung dalam vaksin influenza untuk manusia.
Komposisi
Seluruh virus Influenza tipe A memiliki delapan segmen gen berbeda yang mengkode setidaknya 10 protein virus berbeda. Yang pertama yaitu protein berstruktur di dalam virion yang telah matang/dewasa. Protein dapat dibagi dalam 3 bagian yaitu protein permukaan, yaitu: hemaglutinin (HA), neuraminidase (NA) dan protein saluran membrane ion (M2). Yang kedua yaitu protein internal, yaitu nucleoprotein (NP), protein matriks (M1) dan kompleks polymerase yang terdiri dari Protein Polimerase Dasar 1 (PB1), Protein Polimerase Dasar 2 (PB2), dan Protein Polimerase Asam (PA) [103]. Dua protein tambahan lain yang diproduksi oleh virus Influenza A adalah protein non-struktural, yaitu protein non-struktural 1 (NS1) dan protein non-struktural 2 (NS2), yang juga dikenal sebagai protein ekspor nuklear (NEP) [97]. Protein NS1 dianggap sebagai protein non-struktural sejati yang tidak ditemukan di dalam partikel virus, tetapi diproduksi dalam jumlah banyak di dalam sel hospes [14, 172]. Protein NS2 utamanya ditemukan di dalam sel hospes, namun beberapa protein dapat ditemukan di dalam virion [130]. Beberapa protein aksesori tambahan lainnya telah dideskripsikan sebagai hasil transkripsi dari frame bacaan terbuka (open reading frames), meskipun fungsi sebagian besar dari protein-protein itu masih belum dapat dimengerti dengan baik [177]. Protein PB1-F2, protein dengan 87 asam amino yang ditranskripsikan dari frame bacaan berbeda dari protein PB1, adalah vaktor virulen potensial yang diperkirakan terlibat dalam proses apoptosis sel hospes, namun tidak ditemukan di semua virus influenza tipe A [21]. Protein PA-X, sebuah produk pergeseran frame ribosom telah diketahui dapat memodulasi respons imun pada tikus [51]. Peranan dan pentingnya protein aksesori ini masih dipelajari dan pentingnya peranan mereka dalam patogenesis virus masih belum diketahui.
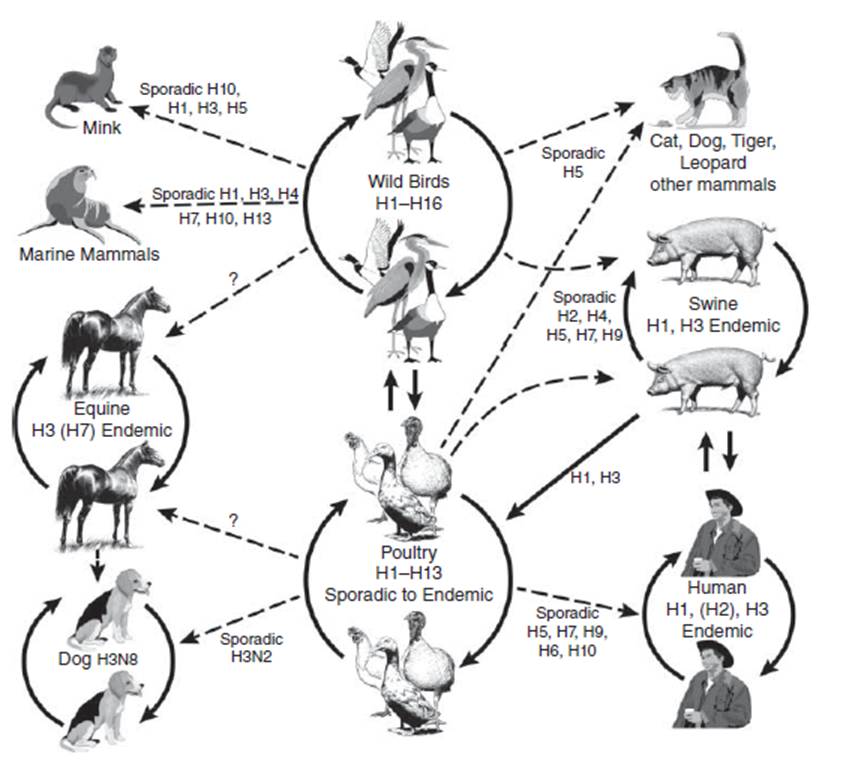
Gambar 1.1 Diagram yang menggambarkan sumber virus dan pergerakan virus atau gen Influenza A di dalam situasi-situasi ekologi dan epidemiologi. (diambil dan diperbaharui dari [160], H = subtipe hemagglutinin, ( ) subtipe yang sebelumnya bersirkulasi secara umum tetap tidak lagi ditemukan peredarannya. Sumber: K. Carter, University of Georgia, dan D. Swayne, USDA/ARS)
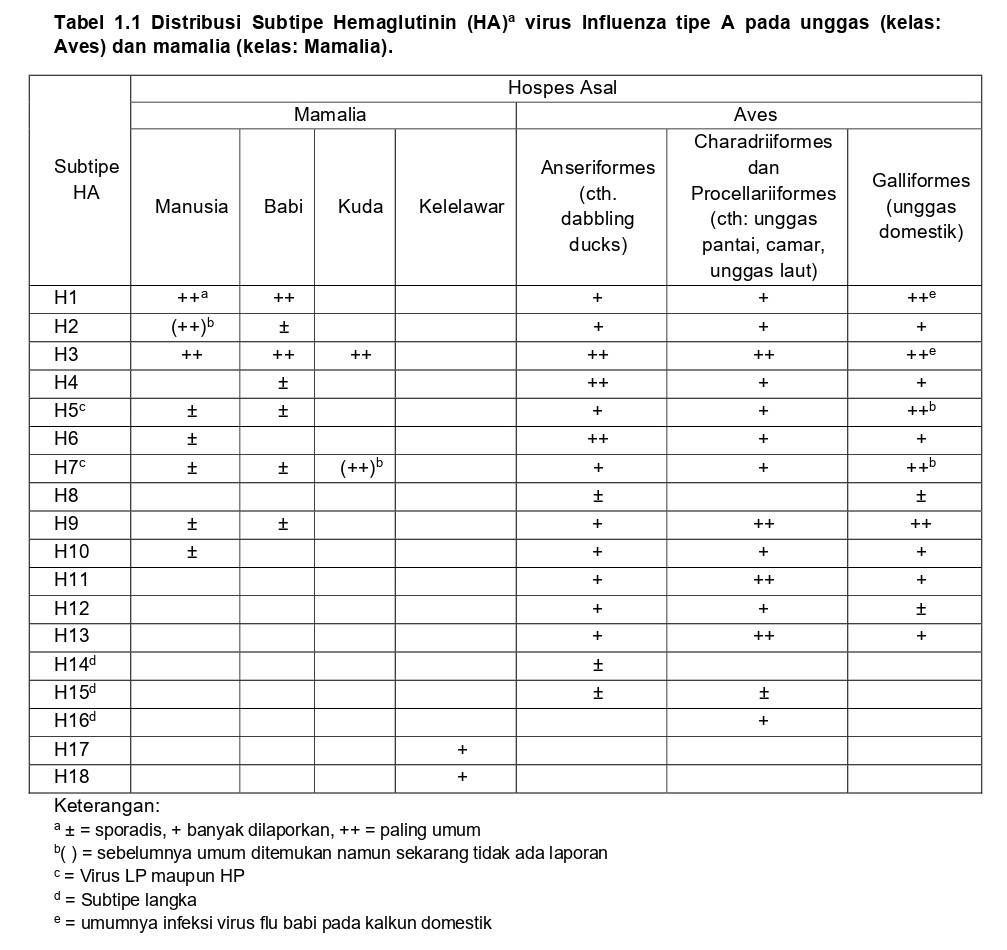
Protein HA dikategorikan ke dalam 18 subtipe, awalnya berdasarkan pada uji hambatan hemaglutinasi (HI), tetapi sekarang telah dikonfirmasi melalui sekuensing dan analisis gen (Tabel 1.1). Subtipe yang berbeda tidak terdistribusi secara secara seragam diantara berbagai spesies unggas dan mamalia, tetapi keanekaragaman yang terbesar ditemukan pada kelas Aves, utamanya pada dua ordo unggas liar; Anseriformes dan Charadriiformes. Distribusi subtipe pada mamalia bersifat lebih terbatas. Beberapa subtipe HA pada mamalia menyebabkan infeksi terbatas yang hanya bersifat endemik maupun sporadik.
Morfologi
Secara morfologi virus Influenza tipe A dapat bervariasi sangat ekstrim, berkisar dari partikel sperikal berdiameter 80-120 nm sampai bentuk benang yang dapat mencapai panjang beberapa mikrometer. Bentuk benang tampaknya mendominasi pada isolat klinis, tetapi setelah dipasase pada kultur sel atau telur ayam berembrio virus seringkali berubah morfologinya menjadi sperikal, hal ini setidaknya ditemukan pada virus manusia [15, 130]. Morfologi virus tampaknya dikontrol oleh protein matriks 1 beserta dua asam amino lain yang juga ditemukan berperan penting pada proses ini [15]. Keseluruhan struktur virus mengandung membrane lipid yang berasal dari sel hospes. Membran lipid ini memiliki tiga protein integral dari virus, yaitu hemagglutinin, neuraminidase dan protein matriks 2. Protein hemaglutinin terdapat dalam bentuk trimer yang muncul sebagai tonjolan duri (spikes) pada membrane lipid, dan merupakan protein permukaan yang paling banyak jumlahnya [25]. Protein neuraminidase terdapat dalam bentuk tetramer dan membentuk struktur globular memanjang keluar dari membrane lipid. Protein M2 adalah protein kecil yang berfungsi sebagai saluran ion yang penting dalam memicu viral uncoating. Protein M1 tampaknya berfungsi sebagi jembatan utama antara membrane lipid dan bagian inti virus yaitu nucleoprotein, RNA virus dan kompleks polymerase.
Propagasi
Virus Influenza tipe A sangat mudah dipropagasi di laboratorium, dan hal ini telah membuatnya lebih banyak dipelajari. Virus Influenza tipe A dari unggas, manusia, babi dan kuda awalnya dipropagasi dalam telur ayam berembrio, dan metode ini masih umum digunakan baik untuk tujuan diagnosis ataupun propagasi virus, terutama untuk produksi vaksin. Saat ini telah banyak pendekatan (emphasis) dilakukan terutama untuk virus influenza tipe A mamalia, yaitu menumbuhkan virus pada kultur sel, baik dalam galur sel primer maupun berkelanjutan, untuk tujuan diagnosis rutin dan produksi vaksin [36, 101, 195]. Galur sel yang umum digunakan untuk isolasi dan propagasi adalah sel fibroblast dari embrio ayam, sel ginjal embrio ayam, sel ginjal Madin-Darby canine, sel Vero, dan lainnya. Untuk virus Avian Influenza (AI), isolasi dan karakterisasi virus umumnya dilakukan pada telur ayam berembrio berumur 9 sampai 11 hari dengan cara inokulasi pada ruang allantois. Telur ayam berembrio memberikan keuntungan tambahan dimana dimungkinkan melakukan replikasi virus LPAI maupun HPAI [41]. Kultur sel embrio ayam primer juga digunakan, tetapi untuk LPAI harus ditambahkan tripsin pada media agar replikasi virus dan pembentukan plak lebih efisien. Penggunaan sistem kultur lain seperti sel ginjal ayam primer dapat dilakukan sebagai alternatif, memungkinkan replikasi dan pembentukan plak virus LPAI tanpa penambahan tripsin, kemungkinan karena sel tersebut memproduksi protease mirip-tripsin seperti yang terlihat pada sel kultur mamalia [62]. Tetapi baru-baru ini penggunaan telur ayam telah terbukti tidak cocok untuk isolasi beberapa virus Influenza tipe A yang berasal dari manusia, babi dan kalkun. Setidaknya sejak 1996 varian H3N2 dari manusia telah diisolasi dalam kultur sel karena tidak lagi tumbuh baik pada telur ayam tanpa adaptasi [195]. Untuk virus-virus ini, isolasi pada kultur sel mamalia lebih dapat diandalkan untuk isolasi primer (awal) [167], meskipun pada salah satu kasus dilakukan inokulasi pada kantong kuning telur yang berhasil mengisolasi virus dibandingkan inokulasi melalui kantong allantois [155]. Virus yang sama yang tidak lagi bereplikasi dengan baik pada telur ayam juga tidak lagi menghemaglutinasi sel darah merah ayam, yang membuat perlunya penggunaan sel darah merah alternatif, seperti sel darah merah kalkun atau marmut [90, 155].
Dimodifikasi dari Swayne,D. E dan M. Pantin-Jackwood. 2008. Pathobiology of avian influenza virus infection in birds and mammals. In: Avian Influenza, D.E. Swayne, ed. Blackwell Publishing: Ames, IA. 87-122.
Nomenklatur
Nomenklatur yang digunakan untuk menggambarkan virus Influenza tipe A telah distandarisasi untuk memberikan nomenklatur yang konsisten dan informatif untuk setiap virus. Kriteria yang digunakan untuk menamai seluruh virus dalam kategori Influenza tipe A terdiri dari; (1) tipe antigen (A, B, C, atau D); (2) hospes hewan asal dimana virus tersebut telah disolasi, tetapi untuk isolat manusia fitur ini dapat dihilangkan atau hanya sekedar diimplikasikan; (3) asal isolat secara geografis, yang dapat berupa kota, negara bagian, provinsi, atau negara; (4) nomor identifikasi unik untuk laboratorium atau referensi lain untuk setiap isolat; (5) tahun yang menunjukkan kapan virus tersebut pertama diisolasi; dan (6) subtipe hemaglutinin dan neuraminidase yang biasanya ditulis di dalam tanda kurung di bagian akhir penamaan. Sebagai contoh, virus influenza yang diisolasi dari kalkun di Missouri dinamai A/turkey/Missouri/24093/1999 (H1N2).
Siklus Hidup Virus
Langkah awal dalam infeksi virus Influenza tipe A adalah perlekatan protein hemagglutinin virus pada reseptor asam sialik pada sel hospes. Perlekatan ini kemudian memacu endositosis. Asam sialik adalah istilah umum yang merujuk pada komponen gula-ujung yang ditemukan pada glikoprotein yang terikat nitrogen dan oksigen. Glikoprotein dapat saja terbuat dari banyak turunan asam neuraminidase. Molekul asal sialik sering diklasifikasikan berdasarkan bagaimana mereka bersambungan dengan gula yang berada pada karbon α-2. Sambungan paling umum ditemukan adalah sambungan α-2, 3 dan α-2, 6 [158]. Sambungan asam sialik yang berbeda ini berakibat pada perbedaan konformasi reseptor protein pada hospes yang selanjutnya akan mempengaruhi perlekatan virus. Protein hemagglutinin, tergantung pada struktur asam aminonya, akan mengikat tipe asam sialik yang berbeda dengan affinitas berbeda pula. Ini yang menentukan apakah virus dapat menginisasi proses infeksi. Virus perlu mengikatkan dirinya cukup kuat pada protein hospes untuk dapat menginisiasi endositosis, dan biasanya memiliki spesifisitas kuat pada salah satu sambungan, baik α-2, 3 ataupun α-2, 6. Spesies hewan berbeda akan memiliki pola dan level ekpresi asam sialik α-2, 3 dan α-2, 6 yang berbeda, yang juga dapat bervariasi antara jaringan satu dan lainnya pada tubuh hewan yang sama. Asam sialik α-2,3 secara dominan terekspresi pada spesies unggas, sedangkan α-2, 6 terekspresi pada manusia. Perbedaan affinitas hemagglutinin diperkirakan menjadi salah satu faktor yang berkontribusi pada batasan (barrier) spesies pada virus influenza tipe A. Bukti-bukti yang ditemukan menyiratkan peranan penting perlekatan tertentu asam sialik. Namun pada beberapa spesies, yaitu manusia, puyuh dan babi, ketiganya mengekpresikan kedua asam sialik (α-2, 3 dan α-2, 6) ini sekaligus, meskipun penyebarannya dalam jaringan berbeda-beda [169, 180]. Distribusi reseptor ini dapat mempengaruhi patogenesis secara langsung, seperti yang telah diperkirakan untuk infeksi H5N1 pada manusia, dimana pneumonia sering sekali ditemukan, dan bukan infeksi saluran pernafasan atas. Patologinya tampak berkorelasi dengan ekpresi asam sialik α-2, 3 pada pneumosit alveolar tipe II pada paru-paru [131]. Faktor tambahan lainnya adalah spesifisitas hemaglutinin terhadap kedua tipe asam sialik tidaklah absolut, dan beberapa virus dapat berikatan dengan asam sialik α-2, 3 maupun α-2, 6 [194]. Pada studi eksperimental pada manusia dan hewan, replikasi kadang terjadi pada banyak virus jika subjek studi diberikan dosis tantangan dalam jumlah cukup banyak [11, 46].
Spesifisitas reseptor hemaglutinin terhadap asam sialik tidaklah absolut, dan dapat berubah sekecil-kecilnya pada substitusi dua asam amino pada posisi 226 dan 228 (penomoran asam amino H30) [26, 179]. Beberapa studi in vivo telah mendokumentasikan sejumlah kasus dimana seleksi perubahan asam amino merefleksikan sistem isolasi atau hospes dimana virus tersebut dipasase [106, 147].
Babi sebelumnya telah diperkirakan sebagai wadah utama percampuran virus influenza manusia dan virus avian influenza karena babi mengekpresikan asam sialik α-2, 3 dan α-2, 6 dalam level yang tinggi pada epitel pernafasannya. Teori yang diajukan adalah bahwa babi dapat terinfeksi virus influenza manusia dan virus avian influenza secara bersamaan, reassortment (penyusunan kembali) dapat terjadi diantara kedua virus ini, menghasilkan virus baru yang dapat menimbulkan strain pandemik [125, 183]. Teori babi sebagai wadah percampuran virus memiliki beberapa dukungan berdasarkan data lapangan, dan virus dengan reassortment yang kompleks telah diisolasi dari babi [56, 176]. Pandemi Avian Influenza H1N1 pada tahun 2009 tampaknya merupakan virus hasil reassortment antara dua virus babi yang berbeda, tetapi identitas hospes dan tempat terjadinya reassortment tidak diketahui [138]. Meskipun begitu, wabah yang terjadi pada manusia dengan virus yang mirip dengan avian influenza (H5N1, H9N2, H7N7, H7N3, dan H7N9), meskipun tidak menjadi pandemi, menunjukkan bahwa kontak dengan unggas terinfeksi dan bukan kontak dengan babi adalah faktor resiko utama terjadinya infeksi [66, 108, 153, 175, 192].
Sekali terjadi perlekatan virus akan mengalami endositosis. Saat endosome menjadi asam proses ini akan memicu aktifnya wilayah fusi pada protein hemaglutinin, dan RNA virus akan dilepaskan ke dalam sitoplasma [146]. Protein M2 memainkan peranan kunci dalam proses pemicu tersebut, karena protein M2 merupakan protein membran integral yang memungkinkan ion H+ memasuki virion, menyebabkan perubahan konformasional pada hemaglutinin pada pH rendah untuk memungkinkan wilayah fusi menjadi aktif [115]. Obat antiviral kelas adamantane beraksi dengan memblok fungsi protein M2, yang mencegah fusi hemaglutinin didalam endosom [43, 157]. Fusi membran virus dan membran endosom, dimediasikan oleh wilayah fusi protein hemaglutinin, memungkinkan pelepasan RNA-polimerase kompleks dari virus ke dalam sitoplasma, dimana ia akan dipindahkan secara aktif ke dalam nucleus dikarenakan oleh adanya sinyal lokalisasi nucleus pada nucleoprotein [96].
RNA virus dengan negative-sense dikopi menjadi mRNA positif-sense oleh polymerase kompleks, yang termasuk didalamnya tiga protein polymerase dan nucleoprotein, dalam nucleus. Virus juga menggunakan protein dalam sel hospes untuk menginisiasi sintesis mRNA, termasuk RNA polymerase II. mRNA membutuhkan primer topi (cap primer) 5’ yang dicuri dari mRNA hospes oleh protein PB2 melalui sebuah proses yang dinamakan perampasan topi (cap snatching) [67]. mRNA virus yang bersifat positif-sense kemudian bermigrasi dari dalam nucleus untuk memulai translasi protein virus didalam sitoplasma dengan mempergunakan sistem di dalam sel hospes. RNA positif-sense juga berfungsi sebagai cetak biru untuk memproduksi RNA negative-sense virus yang nantinya akan dibungkus ke dalam virion.
Dua protein virus, yaitu M1 dan NEP, sangat krusial dalam melancarkan transportasi protein virus masuk dan keluar dari nukleus. Protein M1 juga memerankan peranan penting dalam penyusunan dan pembangunan struktur virion [15]. Proses penyusunan material virus melibatkan tiga protein membran integral, hemaglutinin, neuraminidase dan sejumlah kecil protein M2, memasuki retikulum endoplasma, dimana protein-protein ini dilipat dan diglikosilasi sebelum selanjutnya bergerak menuju membrane plasma apikal [9]. Protein M1 dipercaya berperan kritikal dalam menjembatani membrane protein integral permukaan dengan kompleks ribonukloprotein dan masing-masing dari kedelapan segmen gen virus sebelum pembentukan virion rampung. Kedelapan segmen gen virus ini memiliki area terlindungi, dengan panjang 13 dan 12 nukleotida yang terletak di setiap ujung ke-5’ dan ke-3’ pada setiap segmen, yang memiliki peranan penting dalam pengemasan sinyal. Pengemasan RNA tampaknya bukanlah sebuah proses yang efektif, dan banyak partikel virus yang ditemukan tidak ikut dalam menyusun keseluruhan delapan segmen gen, sehingga proses pengemasan menyisakan partikel-partikel virus sisa yang rusak dalam jumlah banyak. Telah diperkirakan bahwa 90% partikel virus bersifat non-infeksius [29, 31]. Proses pengemasan juga dapat memungkinkan sejumlah segmen gen, terutama gen-gen yang kecil ukurannya, untuk menjadi bagian dari virion. Proses pengemasan yang bervariasi ini bahkan dapat mempengaruhi fenotipe virus, karena telah dihipotesasikan bahwa pada saat beberapa kopian dari gen NS dikemas per virion, peningkatan resistansi virion terhadap produksi interferon dapat terjadi [127].
Pertunasan partikel virus secara efektif dari membrane seluler memerlukan, selain beberapa hal lain, aktivitas enzimatik dari protein neuraminidase untuk menghilangkan asam sialik dari protein hemagglutinin. Hal ini akan mencegah protein untuk berikatan dengan dirinya sendiri dan mencegah agregasi virus pada permukaan sel [89,129]. Pada studi experimental, virus yang mengurangi aktivitas neuraminidase akan mengumpul/beragregasi pada permukaan sel karena partikelnya berikatan satu sama lain, yang dapat mengurangi secara besar-besaran titer efektif dari virus [8]. Hilangnya aktivitas neuraminidase bukan hanya sebuah teori, karena salah satu penanda adaptasi virus avian influenza terhadap unggas adalah dengan ditemukannya keberadaan penghapusan tangkai (stalk deletion) dari protein neuraminidase [88]. Penghapusan tangkai-tangkai ini berakibat pada penurunan aktivitas neuraminidase. Meskipun situs-situs aktifitas neuraminidase tidak terpengaruh oleh penghapusan tangkai, tangkai-tangkai yang pendek diperkirakan mampu untuk mengurasi fleksibilitas protein yang kemudian mengurangi kemampuannya untuk berikatan dengan substrat asam sialik. Virus AIV setidaknya dapat mengimbangi penurunan neuraminidase ini dengan membuat perubahan pada protein hemagglutinin yang mengurangi afinitas pengikatan pada asam sialik, biasanya dengan menambahkan situs-situs glikosilasi baru didekat tempat berikatannya dengan reseptor [91]. Saat ini masih belum dimengerti keuntungan selektif yang didapat dari penghapusan tangkai pada unggas.
Untuk LPAIV, partikel virus yang terlepas tidak bersifat infeksius sebelum protein hemagglutinin terbelah menjadi sub-unit HA1 dan HA2 oleh tripsin atau protease yang mirip tripsin. Peranan pembelahan HA akan didiskusikan lebih detail pada bagian yang membahas tentang patogenesis.
Genetika Virus
Ekologi virus pada unggas liar
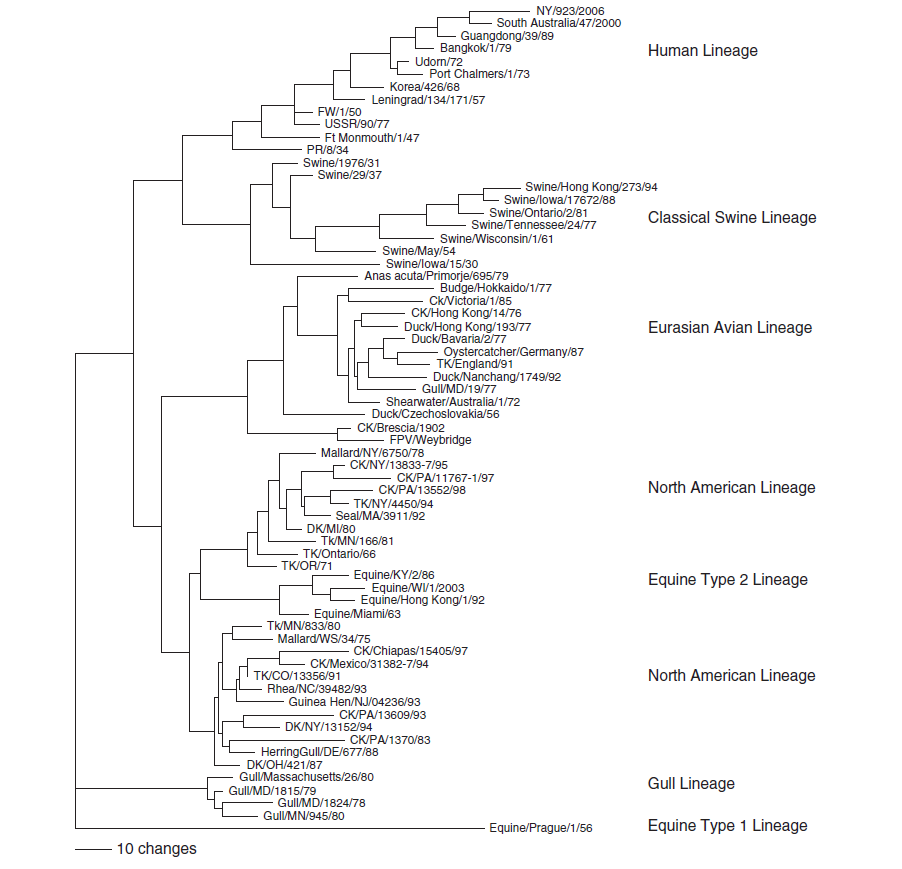
Hospes alami dan reservoir untuk seluruh virus influenza tipe A ditemukan pada unggas liar, terutama unggas air, camar dan burung-burung pantai [58, 133]. Di dalam tubuh hospes alaminya, virus influenza tampaknya berevolusi secara lambat, dengan sebagian besar gen internal tersimpan jauh di dalam susunan asam amino [149]. Glikoprotein permukaan, HA dan NA, lebih bervariasi dalam sekuens asam aminonya, menunjukkan keberagaman yang luas dari gen-gen ini. Untuk kedua protein ini, sejumlah subtipe antigen telah ditandai, dimana antibodi yang terbentuk dari satu subtipe hanya akan menetralisasi virus dengan subtipe tersebut, menunjukkan spesifisitas yang tinggi. Untuk protein HA, 16 subtipe HA AIV telah ditandai (Gambar 1.2), dan untuk protein NA 9 subtipe juga telah ditandai. Pada tataran asam amino, perbedaan antar subtipe sekecil-kecilnya sebesar 20%, akan tetapi subtipe-subtipe paling berbeda dapat mencapai perbedaan sampai 63%. Sekitar 25% dari asam amino disimpan ke dalam 16 subtipe HA [95]. Perbandingan yang serupa juga ditemukan pada subtipe-subtipe NA, dengan persentasi perbedaan asam amino antara 31% dan 61%.
Ketika membandingkan sekuens nukleotida dari beberapa segmen gen yang berasal dari virus AIV unggas liar, termasuk segmen gen di dalam subtpe HA dan NA, ditemukan batasan yang jelas yang terjadi antara virus-virus yang diisolasi di Eropa, Asia, Afrika dan Australia (galur keturunan Eurasia) dan virus-virus yang diisolasi di Amerika (galur keturunan Amerika) [149]. Pada tataran asam amino, untuk protein-protein internal yang lebih tersimpan lagi, perbedaan antara galur keturunan Amerika dan Eurasia telah hilang. Gen-gen HA dan NA yang memiliki keragaman sekuens nukleotida masih memisah pada tataran asam amino, menjadi galur keturunan Eurasia dan Amerika, secara jelas ditunjukkan pada sebagian besar subtipe-subtipe hemagglutinin. Untuk subtipe H7, percabangan galur keturunan lebih lanjut dapat diamati antara jalur keturunan Amerika Utara dan jalur keturunan Amerika Selatan, dan kemudian antara virus dari Australia dan virus yang berasal dari Eropa dan Asia [154]. Perbedaan subtipe-subtipe H7 ini dapat merefleksikan keadaan keterbatasan tersedianya data sekuens, terutama yang berasal dari Amerika Selatan, dimana hanya ada sedikit sekuens AIV yang tersedia. Differensiasi isolat-isolat unggas liar yang dibagi menjadi galur keturunan Dunia Lama dan Dunia Baru mengindikasikan bahwa transfer gen AIV tidak sering terjadi antara kedua area geografis ini. Namun, wabah terkini HPAI H5N8 Eurasia di Amerika Utara pada tahun 2014 menunjukkan bahwa virus-virus ini kadang-kadang dapat berpindah melewati jarak yang jauh [52].
Seiring dengan bertambahnya ketersediaan informasi sekuensing isolat unggas liar dan unggas, aturan yang mendasari galur Amerika dan Eurasia tampaknya mulai menunjukkan beberapa pengecualian. Misalnya, virus influenza A subtipe H2 tampaknya mengikuti aturan galur Amerika dan Eurasia pada isolat unggas dan itik, namun virus-virus Amerika Utara yang berasal dari unggas-unggas laut dan camar lebih berkerabat dekat dengan isolat-isolat Eurasia daripada isolat-isolat H2 lainnya yang berasal dari Amerka Utara [84,124]. Meskipun virus-virus H2 yang berasal dari unggas-unggas laut dan camar tersebut lebih memiliki kesamaan dengan virus-virus dari Eurasia, mereka tetap mengumpul sebagai subgalur keturunan yang unik. Jalur keturunan dari virus-virus yang serupa dengan virus-virus Eurasia dari unggas-unggas laut dan camar juga ditemukan pada virus-virus influenza H6 yang berasal dari Amerika Utara, namun gen internalnya, salah satunya gen matriks dan gen non-struktural, memiliki sekuens yang diperkirakan berasal dari Amerika [143]. Karena itulah, data-data ini kemungkinan mewakili subpopulasi unik dari gen hemagglutinin yang beredar di Amerika Utara, dan bukanlah merupakan bukti adanya perpindahan baru gen-gen virus jalur keturunan Eurasia yang memasuki Amerika.
Kisaran hospes AIV pada burung-burung liar tidak diketahui secara lengkap, tetapi berdasarkan kajian sampling, ada dua ordo dari burung-burung liar yang secara konsisten sering ditemukan tertular, yaitu Anseriformes dan Charadriiformes (Tabel 1.1). Ordo Anseriformes memiliki anggota diantaranya itik dan angsa, tetapi insidensi infeksi tampaknya tertinggi pada kelompok dabbling ducks (subfamily Anatinae)1, yang termasuk di dalamnya diantaranya itik Melewar2, itik Muara3 dan itik Teal4. Insidensi infeksinya tampaknya bersifat musiman, dengan tingkat isolasi tertinggi terjadi pada burung-burung yang muda pada saat musim gugur [145]. Insidensi infeksi yang rendah terjadi pada ordo Charaadriformes, yang mencakup burung-burung pantai dan burung camar. AIV pada unggas luar tampaknya berpindah dengan bebas antara spesies-spesies unggas yang berbeda, dan saat ini tidak dimungkinkan untuk dapat membuat prediksi spesies hospes asal isolat virus berdasarkan sekuens nukleotida. Satu pengecualian pada aturan ini adalah bahwa sebagian besar H13 dan H16 berasal dari burung camar, dan burung camar juga tampaknya memiliki jalur khusus utama yang mencakup beberapa gen-gen internal (Gambar 1.3) [40,152]. Ekologi AIV pada unggas liar akan didiskusikan dengan lebih mendetail pada Bab 8.
Influenza yang berasal dari Kelelawar
Baru-baru ini, dua virus influenza A unik telah teridentifikasi pada beberapa spesies kelelawar, yatu kelelawar berbahu-kuning (yellow-shoulder bats) dan kelelawar muka-rata (flat-faced bats), melalui deteksi molekuler dan sekuens dari sample klinis yang berasal dari Amerika Tengah dan Amerika Selatan. Isolat-isolat dari kelelawar belum bisa didapat melalui kultur sel atau telur berembrio. Sekuens virus menunjukkan kesamaan yang cukup dengan virus influenza sehingga dapat digolongkan dalam genera ini, akan tetapi virus-virus ini juga mempunyai perbedaan unik yang membuat mereka tidak dapat dimasukkan ke dalam klasifikasi virus tipe A tradisional. Virus-virus ini berasal dari dua subtipe baru, H17N10 dan H18N11 [170, 171]. Gen-gen internalnya sepadan dengan gen HA dan NA dari virus influenza manusia dalam sistem genetik terbalik, namun gen-gen HA dan NA memiliki perbedaan struktural yang cukup dimana HA proteinnya menggunakan reseptor yang berbeda dari virus influenza A tipe lainnya, dan gen-gen NA nya tidak memiliki aktivitas neuraminidase yang terukur dan kemungkinan memiliki fungsi yang berbeda [197,199]. Tidaklah mengejutkan bahwa virus-virus mirip influenza telah dideteksi pada kelelawar, karena tingkat kepadatan yang tinggi di dalam koloni kelelawar lebih memungkinkan transmisi virus, tetapi saat ini belum diketahui apakah virus-virus ini menyebabkan penyakit klinis dan sejauh mana persebaran virus pada populasinya.
Epidemiologi virus pada sistem buatan manusia
Virus influenza A bersifat tidak biasa dimana mereka dapat menginfeksi dan bereplikasi pada spesies hospes yang sangat bervariasi secara luas, mencakup ayam, kalkun, babi, kuda, manusia dan spesies unggas dan mamalia lainnya. Akan tetapi, jumlah virus yang diperlukan untuk menginfeksi seekor hospes bisa jadi sangat bervariasi tergantung pada adaptasinya terhadap hospes tersebut. Sifat ini memberikan sedikit tingkat proteksi pada level spesies [141, 173]. Setelah virus beradaptasi dengan hospes baru biasanya akan menjadi lebih susah untuk dapat bereplikasi pada spesies hospes awalnya, seperti misalnya pada burung-burung liar. Jika virus dibiarkan bersirkulasi cukup lama pada hospes yang baru, dia akan menjadi virus yang terspesialisasi untuk hospes tersebut, misalkan menjadi virus yang khusus menyerang manusia, ayam, atau babi. Hal ini berakibat terciptanya galur filogeni yang unik [16, 40]. Virus influenza pada hospes yang baru berubah sangat cepat dan sangat terprediksi yang merupakan sebuah akibat dari tingginya tingkat error pada virus dan tekanan selektif dari hospes [17, 40, 126, 150].
Untuk spesies yang berada dibawah tekanan respon imun yang berasal dari infeksi alami dan/ atau vaksinasi, perubahan gen HA dan NA dapat terjadi dalam tingkat yang lebih cepat lagi [35, 76]. Perubahan yang terjadi pada dua gen ini terkonsentrasi pada situs-situs antigenik secara spesifik. Sebagai contoh, protein H3 pada manusia memiliki lima situs antigenik yang merupakan tempat dimana antibodi netralisasi akan berikatan [182, 184]. Bahkan dengan tingkat pemahaman yang kita punyai sekarang ini, kita tidak dapat memperkirakan perubahan yang akan menimbulkan adaptasi spesies atau membuat virus dapat menghindari respons imun. Jumlah situs asam amino spesifik yang terkait dengan adaptasi spesies terus menerus bertambah. Meskipun ke-delapan gen seluruhnya mungkin saja mempunyai peranan yang penting, gen HA dan PB2 diperkirakan lebih utama dalam membuat perubahan yang penting untuk adaptasi virus dari hospes unggas ke hospes mamalia [93].
Avian Influenza A (AIV) telah menjadi endemik pada sejumlah spesies, diantaranya manusia, babi, kuda dan unggas, dan jika salah satu strain virus influenza bersirkulasi pada spesies tertentu dalam jangka waktu yang panjang (bulanan sampai tahunan), maka virus tersebut akan semakin bersifat spesies-spesifik. Karena itulah, virus manusia biasanya tidak menginfeksi babi, virus kuda tidak menginfeksi kalkun, dan virus unggas tidak menginfeksi manusia. Akan tetapi aturan umum yang menyatakan bahwa virus influenza yang telah beradaptasi pada hospes baru akan tetap berada pada satu spesies dan atau spesies yang memiliki kekerabatan dekat, ternyata memiliki beberapa pengecualian. Sebagai contoh, AIV H1N1 yang klasik berasal dari babi secara rutin melewati batasan spesies dari babi ke kalkun, menyebabkan wabah penyakit yang menimbulkan banyak kerugian [45]. Infeksi sporadis pada manusia oleh beberapa AIV (H5N1, H7N7, H7N3, H7N7 dan H9N2) dari unggas telah teramati, dan karena itu AIV memiliki resiko sebagai patogen zoonosis, meskipun resiko tersebut tergolong rendah [66, 108, 156, 175, 192]. Beberapa studi tantang eksperimental pada manusia telah dilakukan menggunakan virus AIV, tetapi secara umum virus bereplikasi dengan kualitas rendah dan hanya menimbulkan penyakit ringan atau tidak bergejala sama sekali [11]. Tidak dimengerti apakah semua subtipe HA dan NA memiliki kemampuan yang sama dalam menginfeksi manusia ataupun spesies lainnya. Saat ini hanya terdapat segelintir subtipe yang telah menjadi endemik pada manusia (H2, H2, N1, dan N2) [190].
Pergerakan AIV dari burung-burung liar ke spesies unggas-unggas domestik tidak jarang terjadi, namun sangat jarang berakibat menjadikannya endemik pada unggas. Beberapa rute pertemuan antara virus dari unggas-unggas liar dan unggas telah terdokumentasikan atau telah diduga sebagai asal muasal terjadinya wabah. Kontak langsung dengan unggas-unggas liar diduga merupakan metode utama perpindahan ini. Beberapa kasus yang terdokumentasi sangat baik tentang hal ini adalah kasus yang terjadi pada peternakan kalkun komersial di Minnesota, dimana sejumlah wabah Avian Influenza teramati pada tahun 1980an dan pada awal 1990an [42]. Virus AIV dengan subtipe HA dan NA yang beragam telah diisolasi dari kalkun selama beberapa wabah, dan biasanya ini terjadi pada saat itik liar bermigrasi saat periode breeding musim panas mereka. Selama musim migrasi itik liar, kalkun diumbar di luar kandang dan unggas liar bisa saja terbang di atas mereka atau mendarat di tempat kalkun diumbar. Pada tahun 1990an sistem manajemen diubah, dimana kalkun tidak lagi diumbar dan ditempatkan dalam kandang tertutup sepanjang hidup mereka, dan insidensi AIV menurun menjadi sangat rendah [164]. Pembatasan kontak antara unggas dan unggas liar melalui pemeliharaan kandang tertutup dan pendekatan biosekuriti lainnya telah memberikan kesempatan untuk mengurangi resiko masuknya virus AIV dari burung-burung liar.

Gambar 1.2 Pohon filogeni dari ke-17 subtipe hemaglutinin (HA). Sekuens asam amino lengkap dari isolat yang mewakili keseluruhan 16 subtipe HA avian dan H17 subtipe kelelawar juga dimasukkan dalam daftar ini, dengan perwakilan dari isolat asal Amerika Utara dan Eurasia jika tersedia. Pohon filogeni ini diakarkan menggunakan program Influenza Research Database PhyML, versi 3.0 [144].
Sumber lain masuknya AIV ke dalam populasi unggas adalah melalui sistem perdagangan unggas hidup (Live Poultry Market /LPM), yang ditemukan pada banyak negara di dunia, termasuk Amerika Serikat. LPM biasanya memberikan banyak jenis unggas yang nantinya akan disembelih dan digunakan untuk konsumsi manusia. Pada beberapa negara dimana fasilitas pembekuan daging tidak tersedia, sistem ini memungkinkan produk untuk tetap segar sebelum dijual. Pada beberapa negara lainnya, seperti Amerika Serikat dan Hong Kong, sistem LPM memiliki pasar untuk para pembeli yang menginginkan pilihan daging segar dari jenis unggas yang ingin dikonsumsi secara spesifik dengan pilihan harga premium dibandingkan dengan membeli daging unggas dingin atau beku dari supermarket. Akan tetapi sistem pemasaran ini memberikan sebuah lingkungan yang ideal untuk pemasukan virus dan mempertahankan virus ini tetap ada di dalam populasi unggas [70, 150]. Sebuah skenario yang biasa terjadi adalah ketika unggas air domestik, terutama itik, dibesarkan di kolam-kolam air dimana itik liar dan unggas-unggas liar lainnya umum ditemukan [10]. Keadaan ini menciptakan resiko tinggi terjadinya infeksi pada itik domestik, yang kemudian akan ditularkan ke dalam sistem LPM saat terjadi kontak dekat dengan unggas lain, seperti ayam, unggas puyuh dan unggas-unggas gallinaseus lainnya. Suplai unggas-unggas baru yang tidak memiliki kekebalan terhadap AIV secara berkelanjutan masuk ke dalam system LPM, dan keadaan ini memberikan kesempatan untuk virus beradaptasi pada hospes baru yaitu ayam dan spesies unggas lainnya. Saat AIV telah bercokol di dalam system LPM, system ini akhirnya menjadi sumber berkelanjutan masuknya infeksi kembali ke dalam peternakan komersial. Salah satu contohnya adalah AIV H7N2 yang mulai bersirkulasi di area timur laut Amerika pada tahun 1994 dan dihubungkan dengan setidaknya lima wabah berbeda pada industri peternakan unggas di tujuh negara bagian sebelum akhirnya berhasil dieradikasi [142]. Kecemasan tentang peranan LPM dalam masuknya virus AIV telah berakibat pemerintah pada Hong Kong melarang jual beli itik dan angsa hidup di pasar-pasar, melakukan program surveillans secara komprehensif, dan memperketat persyaratan sanitasi produk [70]. Burung puyuh juga telah terimplikasi sebagai spesies yang sangat rentan yang mungkin memiliki peranan penting dalam transmisi virus di dalam sistem pemasaran [85, 106]. Perubahan manajemen dan biosekuriti telah terbukti efektif dalam mengurangi insidensi infeksi unggas dalam sistem pemasaran.
Resiko tambahan masuknya virus ke dalam sebuah peternakan adalah melalui air minum. Hal ini biasanya terjadi saat sumber air permukaan, seperti danau dan sungai, digunakan sebagai sumber air minum unggas atau untuk aktivitas lainnya di sekitar peternakan. Jika air minum tidak dimurnikan dengan baik, maka virus AIV dari unggas-unggas liar dapat masuk ke dalam kawanan unggas melalui air tersebut. Penggunaan air minum mentah telah diperkirakan sebagai sumber munculnya wabah AI di Amerika Serikat, Australia dan Chile [47, 132, 154].
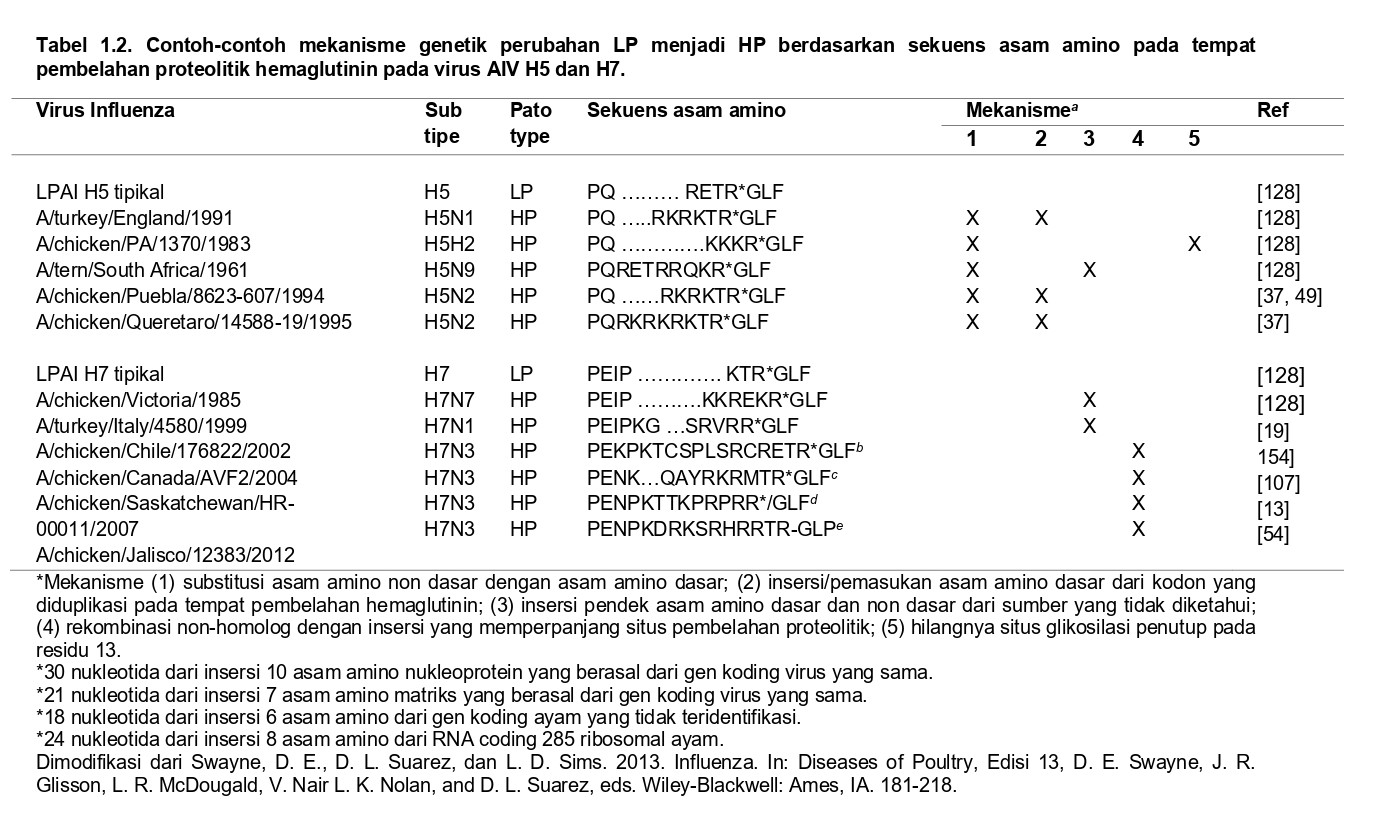
Gambar 1.3 Pohon filogeni dari gen matriks. Pohon ini disusun berdasarkan sekuens nukleotida komplit dari isolat-isolat yang mewakili grup-grup utama dari virus influenza A. Pohon ini diakarkan kepada equine/Prague/1/56, yang merupakan virus influenza A yang paling beragam. CK=Chicken/Ayam, TK=Turkeys/Kalkun. Singkatan standar yang terdiri dari dua huruf digunakan untuk asal negara bagian untuk isolat-isolat yang berasal dari Amerika Serikat.
Setidaknya ada satu lagi sumber transmisi virus AIV yang umum terjadi pada kalkun adalah kontak dengan babi yang terinfeksi virus flu babi (swine influenza virus / SIV). Kalkun rentan terhadap SIV dan jika peternakan kalkun dekat dengan peternakan babi maka dapat menjadi faktor resiko masuknya virus SIV. Infeksi SIV H1N1 klasik, SIV H1N2 dan H3N2 yang baru saja ditemukan, dan virus PH1N1 pada kalkun telah dilaporkan [45, 105, 155, 1919]. Flu babi memiliki sejarah yang unik dan kompleks yang memiliki beberapa kesamaan dengan penyakit pada unggas, tetapi juga memiliki beberapa perbedaan yang penting. Gen virus flu babi juga diperkirakan berasal dari unggas-unggas liar tetapi terdeteksinya gen virus AI pada virus flu babi baik secara in toto maupun sebagai hasil reassortment dengan virus SIV yang endemik relatif jarang ditemukan.
Strain virus flu babi di Amerika Utara dan Eropa cukup terpisah sebelum terjadinya pandemik H1N1 (pH1N1) pada manusia pada tahun 2009. Virus-virus pH1N1 ini mampu menginfeksi bukan hanya manusia, tetapi juga babi, kalkun, ferret/musang dan menyebabkan kasus sporadis pada beberapa spesies lain [105, 178]. Sumber infeksi flu babi di Amerika Utara dihubungkan dengan pandemi Flu Spanyol H1N1 pada tahun 1918. Virus tersebut memisahkan diri dari cabang virus isolat manusia dan relatif stabil selama hampir 80 tahun, dan telah diketahui dengan sebutan “classical swine influenza”. Kemudian pada tahun 1998 virus flu babi baru muncul di Amerika Serikat. Virus ini memiliki kaset gen internal yang unik, tersusun dari gen-gen virus AIV babi, manusia, unggas dan gen H3 dan N2 yang mirip virus influenza manusia [181, 198]. Kaset gen internal triple reassorment (TRIG) memungkinkan terjadinya banyak reassortment pada subtipe HA dan NA yang berbeda. Kaset TRIG mencakup banyak gen yang membentuk dasar dari pandemi H1N1 pada manusia pada tahun 2009 [138]. Sejak 1999, beberapa varian antigenik dan kejadian reassortment pada virus manusia telah menciptakan koleksi virus yang terus berubah di Amerika Utara [5].
Virus flu babi klasik bersirkulasi di Eropa selama bertahun-tahun, tetapi digantikan oleh virus AIV yang berasal dari unggas pada tahun 1979 [68]. Virus flu babi yang mirip dengan flu asal-unggas menjalani assortment dengan virus H3N2 pada manusia pada tahun 1984 untuk menciptakan jalur keturunan yang stabil. Banyak virus reasortan lain yang dengan asal yang berbeda-beda telah terdeteksi pula dan virus H1N2 adalah virus yang umumnya paling banyak ditemukan [72]. Virus pH1N1 manusia telah masuk ke dalam peredaran pada tahun 2009 dan saat ini gen-gen virus H1N1 mirip virus unggas, gen H3N2 dan H1N2 mirip virus manusia dengan kaset gen internal yang berbeda, dan gen pH1N1 telah bersirkulasi pada populasi babi di Eropa [178].
Meskipun surveilans pada populasi babi di Amerika Utara dan Eropa sangat jauh dari komprehensif untuk virus flu babi, telah tersedia isolat-isolat yang cukup merepresentasikan dan mendokumentasikan varian-varian virus tersebut. Surveilans di Asia lebih bersifat sporadic, akan tetapi masih tetap dapat mendokumentasikan variasi virus yang berdedar pada populasi babi, seperti diantaranya: classic SIV, SIV Eropa yang mirip virus dari unggas, virus influenza manusia dan virus-virus H1N1, H3N2 dan H1N2 yang tidak ditemukan di Eropa dan Amerika Utara. Tingginya kepadatan populasi babi dan importasi babi ke dalam salah satu wilayah memberikan sebuah tempat percampuran virus-virus unik dari seluruh dunia [178]. Surveillance virus babi hampir-hampir tidak dilakukan di Australia, Afrika dan Amerika Selatan sebelum 2009, saat dimana pandemi H1N1 pada manusia muncul ke permukaan. Penelitian-penelitian telah mendokumentasikan tentang babi-babi yang terinfeksi virus pH1N1 di ketiga benua tersebut, dan di Australia, virus infeksi ini baru saja terdokumentasikan kembali setelah lama berstatus bebas dari SIV [28, 94]. Beberapa subtipe virus teridentifikasikan di Argentina, salah satu diantaranya virus-virus unik yang berasal dari manusia, demikian juga dengan pH1N1 [110].
Kemunculan pH1N1 memberikan sebuah dorongan yang dibutuhkan untuk dapat meningkatkan surveilans pada babi karena pH1N1 memiliki asal muasal yang jelas yaitu dari SIV, akan tetapi akan dan dimana jalur keturunan virus ini bakal muncul masih belum diketahui. Kemunculan virus-virus baru pada babi mengindikasikan bahwa gen-gen virus bisa saja berasal dari sumber-sumber yang bervariasi, termasuk juga dari unggas dan manusia. Pembatasan pergerakan babi telah menyebabkan jalur keturunan virus terbentuk di Eropa dan Amerika Utara, meskipun terdapat overlap virus-virus di Asia. Bukti-bukti infeksi pada babi oleh virus-virus IAV yang berasal dari unggas, baik berasal dari unggas-unggas liar atau dari unggas domestik terus-menerus dilaporkan, dan hal ini memberikan ancaman munculnya virus-virus baru yang berimplikasi terhadap kesehatan hewan dan manusia [44, 55, 83].
Virus Influenza pada Kuda dan Anjing
Hanya ada dua galur virus influenza pada kuda (EIV) yang telah dilaporkan bersifat endemik di dalam populasi kuda. Subtipe asal yang terdeteksi berasal dari virus H7N7 yang pertama kali diisolasi pada tahun 1956. Berdasarkan sekuensing galur EIV H7N7 merupakan cabang dari virus influenza lain. Virus ini telah berada di dalam populasi dalam waktu yang lama [189]. Masuknya virus H3N8 pada tahun 1963 berakibat kepunahan dari galur virus H7N7. Galur H3N8 telah menginfeksi kuda di seluruh dunia, kemungkinan sebagai akibat adalah pergerakan kuda secara internasional untuk keperluan pacuan kuda atau even-even terkait kuda lainnya. Mirip dengan virus influenza pada manusia yang juga memiliki sebaran di seluruh dunia, virus H3N8 telah terus berevolusi menjadi subgalur yang unik, meskipun hanya ada beberapa subgalur, kemungkinan dikarenakan virus yang lebih fit akan mengalahkan virus yang kurang kompeten. Saat ini, dua clade yang berasal dari subgalur Florida merupakan strain yang dominan [24, 39]. Terlihat pada salah satu contoh paling jelas adanya lompatan (jumping) virus influenza melewati batasan (barrier) spesies, yaitu virus EIV clade 1 H3N8 melakukan lompatan ke anjing, kemungkinan di Florida, yang berakibat terbentuknya galur virus influenza anjing yang unik [24]. Sebuah studi yang baru telah menunjukkan bahwa virus yang telah beradaptasi di tubuh anjing telah mengurangi virulensinya terhadap kuda [119]. Even unik yang kedua juga telah dilaporkan, dimana H3N8 melompat dari kuda ke anjing yang terjadi di Australia selama epidemi yang terjadi di negara itu pada tahun 2007 [63].
Penyakit Klinis pada Unggas
Penampakan di lapangan
Virus influenza pada unggas, terutama pada ayam dan kalkun bisa saja tanpa gejala, akan tetapi juga sering menyebabkan kerugian produksi dan penyakit klinis yang berkisar antara gejala ringan sampai berat pada flok-flok yang tertular. Virus-virus ini secara umum dapat dibagi menjadi virus-virus yang menyebabkan infeksi mukosa saluran pernafasan dan atau saluran pencernaan, dan virus-virus yang juga menyebabkan infeksi sistemik. Virus-virus yang menyebabkan infeksi mukosa biasanya disebut dengan LPAIV (Low Pathogenic Avian Influenza Virus), dan umumnya virus-virus ini tidak menimbulkan mortalitas yang tinggi pada flok yang terkena. Virus-virus yang menyebabkan infeksi sistemik biasanya menyebabkan angka mortalitas yang tinggi dan disebut dengan HPAIV (Highly Patogenic Avian Influenza Virus) yang secara historis sebelumnya disebut virus sampar unggas [64].
LPAIV dapat menyebabkan infeksi asimptomatik tetapi umumnya menyebabkan penyakit klinis ringan sampai berat. Penurunan konsumsi pakan dan air minum adalah sebuah indikasi lain terjadinya penularan yang akan teramati jika pencatatan jumlah konsumsi dilakukan dengan cermat. Untuk flok layer ataupun breeder, penurunan produksi telur juga dapat teramati. Penurunan produksi telur ini bisa saja berat, menyebabkan produksi flok tidak pernah kembali ke kondisi berproduksi penuh, seperti yang biasa terlihat pada breeder kalkun yang terinfeksi dengan virus yang mirip dengan virus influenza babi [45,92]. Dalam flok yang besar, peningkatan sejumlah kecil kematian dapat teramati saat virus menyebar ke seluruh flok. Infeksi LPAIV berkontribusi pada peningkatan kematian, karena itulah pengujian diagnostik terhadap kematian harian disebut sebagai cara sensitif dalam mengindentifikasi infeksi LPAIV [3, 151]. Pada beberapa situasi, infeksi LPAIV dapat berakibat kematian yang tinggi, secara umum diasosiasikan dengan keberadaan infeksi patogen atau infeksi sekunder dan/atau kondisi lingkungan yang buruk [7]. Pada kejadian yang jarang terjadi, LPAIV dapat menyebabkan lesi spesifik pada organ dalam, baik melalui infeksi langsung maupun melalui sebab-sebab tidak langsung [200].
Penyakit dan lesi yang disebabkan oleh infeksi AIV pada itik domestik akan didiskusikan secara lebih detail pada Bab 14, dan pada Bab yang membicarakan patobiologi dari infeksi virus avian influenza pada unggas dan mamalia yang terdapat pada edisi terdahulu dari buku ini [160]. Di dalam buku edisi ini, topik terkait dapat ditemukan pada Bab 5: penyakit dan lesi disebabkan infeksi AIV pada manusia, Bab 16: pada babi, Bab 20: pada kuda, Bab 22: pada anjing, Bab 23: pada beberapa mamalia, dan Bab 24: pada hewan laboratorium.
Karakterisitik molekuler dan biologis dari virus LPAIV dan HPAIV
LPAIV dapat berupa subtipe-subtipe yang berbeda komponen neuraminidase dan hemaglutininnya. Sedangkan HPAIV terbatas pada subtipe H5 dan H7 dengan alasan yang belum diketahui, meskipun sebagian besar virus influenza H5 dan H7 memiliki patogenitas rendah. Sangat jarang ditemukan LPAIV dari H5 dan H7 ini bermutasi menjadi HPAIV. Dipercayai bahwa secara umum HPAIV berasal dari virus H5 dan H7 LPAIV yang telah bersirkulasi di unggas dalam waktu yang lama. Sebagai contoh, LPAIV yang bersirkulasi selama beberapa bulan sampai tahunan pada flok unggas pada kasus wabah virus H5 di Pennsylvania pada tahun 1983 dan Meksiko pada tahun 1994, dan wabah virus H7 di Italia pada tahun 1999, sebelum virus tersebut bermutasi menjadi HPAI [49, 59, 196]. Tekanan seleksi virus untuk berubah dari LPAIV menjadi HPAIV tidak diketahui saat ini, tetapi replikasi virus yang terjadi di dalam tubuh unggas, seperti ayam, kalkun dan unggas puyuh diperkirakan merupakan bagian kritis dari proses ini. HPAI dipercaya secara normal tidak beredar pada hospes reservoir yaitu unggas-unggas liar [121]. Akan tetapi pada empat kejadian yang terpisah, HPAI telah terdeteksi pada unggas-unggas liar. Wabah yang pertama terjadi pada populasi unggas dara laut (tern) di Afrika Selatan pada tahun 1961, yang tidak berhubungan dengan sumber unggas [12]. Tiga wabah H5 HPAI yang merebak secara luas pada unggas-unggas liar telah dilaporkan dalam kurun waktu 10 tahun yang kesemuanya diasosiasikan dengan wabah pada unggas. Kejadian awal spillover (spillover) clade 2.2 H5N1 pada tahun 2005 berakibat pada kejadian-kejadian kematian pada berbagai spesies unggas liar. Virus berpindah melalui unggas-unggas liar sampai akhirnya mencapai sebagian besar benua Eropa dan beberapa negara di Afrika. Galur virus ini tidak bertahan secara permanen pada unggas liar [82]. Kejadian spillover kedua yang terekam adalah dari clade 2.3.2.1 virus H5N1 yang pertama kali terdeteksi pada tahun 2007 [139]. Virus ini pertama kali terdeteksi utamanya di Asia Timur, tetapi akhirnya menyebar ke negara-negara Eropa Timur dan Asia Selatan pada tahun 2010 dan 2011, dan menjadi terintegrasi ke dalam populasi unggas di Bangladesh. Isolat dari unggas-unggas liar biasanya diambil dari unggas yang mati ataupun sakit, tetapi tidak berasosiasi dengan kejadian kematian yang besar pada unggas-unggas liar yang menandai kejadian awal masuknya virus clade 2.2 ke unggas liar. Pengujian eksperimental menunjukkan adanya virulensi yang tinggi pada ayam, akan tetapi mortalitas yang bervariasi pada spesies-spesies itik yang berbeda [22, 33, 53, 99]. Masih belum jelas apakah galur ini bertahan pada unggas-unggas liar.
Wabah yang menyebabkan angka morbiditas tinggi (epornitik) yang terjadi pada unggas-unggas liar dideteksi telah terjadi pada akhir tahun 2013 dan telah menyebar dari Asia Timur ke Eropa dan Amerika Utara. Virus penyebab wabah ini terdiri dari beberapa reassortan, virus N8 adalah penyebab dominan, tetapi reasortan N2 dan N1 juga telah terdeteksi. Gen hemaglutininnya diklasifikasikan sebagai clade 2.3.4.4. Virus ini juga tidak diasosiasikan dengan angka mortalitas yang tinggi pada unggas-unggas liar dan tampaknya memiliki virulensi rendah pada ayam jika dibandingkan dengan virus H5N1 yang sudah dikenal [32, 140].
Patobiologi seluler dan pembelahan hemaglutinin
Karakteristik virulensi utama yang memisahkan LPAIV dan HPAIV pada ayam dan unggas-unggas galinacea lainnya adalah kemampuan protein hemagglutinin untuk dibelah oleh protease yang terdapat di dalam sebagian besar sel hopses. Virus influenza harus membelah protein HA, yang diproduksi sebagai polipeptida tunggal, menjadi 2 subunit, yaitu HA1 dan HA2 sebelum bisa menginfeksi. Pembelahan ini diperlukan agar area fusi tetap aktif selama langkah pengelupasan lapisan di dalam proses replikasi virus. Normalnya protease tripsin atau yang mirip tripsin (plasmin, faktor pembekuan darah yang mirip protease, Clara triptase, protease bakterial) membelah protein hemagglutinin dengan mengenali arginin tunggal yang berada di lingkungan ekstraseluler [41, 62, 65, 73]. Distribusi LPAIV di dalam tubuh hospes dipercaya dipengaruhi oleh ketersediaan protease mirip tripsin ini secara lokal pada salurah respirasi dan pencernaan [65]. Protease lain juga dapat membelah virus influenza, dan pada embrio ayam enzim yang mirip prothrombin yang mirip dengan faktor X pembekuan darah dipercaya sebagai agen yang melakukan pembelahan ini [41]. Akan tetapi jika beberapa asam amino dasar (lisin dan arginin) terdapat pada tempat pembelahan HA, khususnya dengan sisipan beberapa asam amino dasar, tempat pembelahan ini menjadi dapat diakses oleh furin atau protease-protease lain yang terdapat dimana saja di dalam sebagian besar sel di tubuh hospes [148]. Protein HA yang berasal dari HPAIV membelah selama langkah penyusunan kembali di dalam proses replikasi virus, dan karena itulah ia bersifat infeksius ketika dilepaskan dari sel [146, 148]. Kejadian ini memungkinankan virus HPAIV untuk dapat mengembangkan kemampuannya untuk bereplikasi di dalam banyak sel dengan tipe berbeda, yaitu dalam beragam tipe sel yang ditemukan di dalam otak, jantung, otot-otot skeletal, dan pankreas. Kerusakan kritis pada organ-organ vital dan atau sel-sel endothelial yang melapisi dinding pembuluh darah dapat menyebabkan gejala-gejala penyakit yang seringkali berakhir dengan kematian hospes unggas tersebut [111, 159]. Gen virus lainnya juga berperan penting dalam menentukan virulensi virus, tetapi tempat pembelahan hemagglutinin sejauh ini merupakan ciri virulensi yang paling penting pada unggas-unggas gallinaseus [81, 123].
Dampak jenis hospes dan strain virus terhadap patogenitas virus
Penotipe HPAIV sesuai dengan namanya menyebabkan tingginya angka mortalitas pada ayam SPF umur 4 sampai 6 minggu [188], tetapi hanya karena virus ini bersifat sangat pathogen pada ayam hal ini bukan berarti dapat dijadikan sebagai prediktor jalannya penyakit pada spesies lainnya. Beberapa kajian telah mengakarakterisasikan patogenitas beberapa isolat tunggal terhadap sejumlah spesies berbeda dalam sebuah uji tantang eksperimental. Salah satu kajian berantai dengan cakupan yang luas memeriksa virus H5N1 HPAI yang diisolasi tahun 1997 asal ayam dari Hong Kong yang digunakan sebagai inokulum eksperimental untuk spesies unggas yang bervariasi. Strain Hong Kong 97 menyebabkan mortalitas tinggi pada semua spesies gallinaseus dalam penelitian, yaitu ayam, kalkun, burung puyuh dan burung pegar, meskipun rata-rata waktu kematian tercatat berbeda-beda untuk setiap spesies [111]. Sebagian besar spesies yang dites menunjukkan gejala klinis yang tidak begitu berat atau untuk beberapa kasus tidak menimbulkan gejala klinis, meskipun sebagian besarnya diinfeksikan berdasarkan bisa tidaknya dilakukan reisolasi virus dari unggas yang di-uji tantang [112-114]. Prediksi virulensi untuk spesies selain spesies gallinaseus tidak dapat dilakukan untuk unggas-unggas dari ordo lain. Sebagai contoh, beberapa angsa yang di-uji tantang menunjukkan gejala syaraf dan lesi yang berkorelasi dengan tempat replikasi virus di otak [112], sedangkan itik yang berasal dari ordo yang sama dengan angsa, yaitu Anseriformes, hanya menunjukkan infeksi terbatas pada saluran pernafasan, dan tidak menujukkan bukti-bukti adanya penyakit/ tidak bergejala [112]. Tampaknya jelas bahwa virulensi yang berhubungan dengan tempat pembelahan hemagglutinin bukanlah satu-satunya faktor yang menentukan virulensi pada spesies lainnya. Hal ini telah jelas terlihat pada itik yang terinfeksi dengan virus H5N1 belakangan ini. Pada hewan model itik Peking umur 2 minggu, virus-virus yang merupakan isolat awal dari tahun 1997 sampai 2001 dapat menginfeksi tetapi tidak menyebabkan morbiditas dan mortalitas. Akan tetapi, dimulai dengan beberapa isolat yang diisolasi dari tahun 2002 terlihat adanya peningkatan mortalitas, dengan isolat yang lebih baru memperlihatkan 100% mortalitas [104, 161]. Virus H5N1 Asia semuanya memiliki gen H5 dari galur yang sama dan memiliki sekuens tempat pembelahan H5 yang identik atau hampir identik dengan sisipan beberapa asam amino dasar dan tetap bersifat sangat patogenik pada ayam. Akan tetapi, gen internal pada virus-virus ini bervariasi, dan dipercaya bahwa perbedaan gen internal ini menyebabkan perbedaan virulensi [78].
Untuk spesies-spesies mamalia, termasuk babi dan manusia yang secara alami terinfeksi HPAIV, penyakit klinis berat yang muncul diasosiasikan dengan pneumonia atipikal berat yang merefleksikan tempat replikasi virus yang utamanya berada pada saluran respirasi, dan replikasi secara sistemik tidak umum terobservasi. Spesies mamalia lainnya yaitu musang, kucing dan anjing mungkin saja mengalami persebaran virus secara sistemik yang berkontribusi pada tingginya tingkat kematian untuk beberapa strain HPAIV [69]. Patogenesis HPAIV sulit untuk dikarakterisasikan untuk semua spesies, dan seiring dengan perubahan pada virus, presentasi gejala klinis yang muncul juga sering berubah.
Perubahan Hemaglutinin yang diasosiasikan dengan patogenitas yang tinggi
Tempat pembelahan hemaglutinin tetap merupakan prediktor terbaik meskipun tidak sempurna untuk memprediksi virulensi virus pada ayam dan spesies unggas-unggas gallinaseus lainnya. Seperti yang sudah disebutkan sebelumnya, keberadaan sejumlah asam amino dasar pada tempat pembelahan HA1 dan H12 berkorelasi dengan virulensi [122]. Hanya subtipe H5 dan H7 yang saat ini diketahui memiliki penotipe H5N1, untuk alasan yang belum terlalu jelas. Perbandingan sekuensing menunjukkan bahwa subtipe H5 dan H7 sangat berbeda satu sama lainnya. Meskipun baik protein H5 dan H7 menunjukkan prinsip dasar bahwa tempat pembelahan, antara arginin dan glisin dan sejumlah asam amino dasar yang berada pada tempat pembelahan berakibat pada munculnya penotipe HPAIV, terdapat perbedaan yang jauh antar subtipe. Sekuens tempat pembelahan yang khas pada virus LPAIV H5 dan H7 adalah berbeda pada burung-burung liar [121]. Virus-virus H5 umumnya memiliki sekuens QRETR/G dengan posisi arginin pada -1 dan 13. Perubahan virulensi virus H5 dapat terjadi dengan adanya subsitusi atau pergantian asam amino non dasar kepada asam amino dasar atau dengan masuknya amino non dasar dan asam amino dasar pada tempat pembelahan (Tabel 1.2). Virus chicken/Scotland/5/H5N1 memiliki empat asam amino dasar pada tempat pembelahan RKKR/G [27], kemungkinan melalui tempat-tempat subsitusi yang berakibat pada munculnya fenotipe HPAI. Secara umum, asam amino tambahan dimasukkan pada tempat-tempat pembelahan, dan ada dua, tiga atau empat asam amino tambahan telah terobservasi. Sebagai contoh, virus H5N1 chicken/Hong Kong/97 memiliki sekuens QRERRRKKR/G [153]. Mekanisme masuknya asam amino ini tidak jelas, tetapi kejadian duplikasi tempaknya terjadi pada beberapa virus HPAIV H5 [109]. Bagian lain dari protein hemaglutinin juga dapat memainkan peranan dalam menentukan fenotipe virus. Sebagai contoh adalah ada atau tidak adanya wadah glikosilasi pada posisi 10-12 pada protein HA1. Pada tahun 1983, virus LPAI H5N2 chicken/Pennsylvania/1/1983 telah diisolasi, memiliki empat asam amino dasar, QRKKR/G pada tempat pembelahan. Enam bulan kemudian, HPAIV merebak di Pennsylvania, virus yang diisolasi dari wabah adalah chicken/Pennsylvania/1370/83 yang memiliki tempat pembelahan HA yang sama akan tetapi virus ini telah kehilangan tempat glikosilasi pada posisi 10-12 pada protein HA1. Tempat glikosilasi ini secara struktural sangat dekat dengan tempat pembelahan HA dan dipercayai bahwa hilangnya komponen gula memungkinkan akses yang lebih besar terhadap tempat pembelahan, mengakibatkan tingginya aksesibilitas enzim protease untuk mencapai tempat pembelahan yang dapat merubah fenotipe virus [59]. Dalam percobaan secara eksperimental, fakta ini dan juga tempat glikosilasi telah ditunjukkan sebagai faktor penting yang mempengaruhi virulensi [50].
Perubahan virus H7 virus LPAIV menjadi HPAIV tampaknya memiliki beberapa perbedaan yang penting. Pertama, seluruh virus H7 HPAI memiliki insersi 2 sampai 10 tambahan pada situs pembelahan. Mekanisme insersi semacam ini juga tampaknya berbeda pada banyak kasus. Meskipun kejadian duplikasi sepertinya terjadi pada beberapa virus, pada beberapa kasus baru-baru ini rekombinasi non-homolog tampaknya merupakan metode terjadinya insersi. Pada wabah di Chile pada tahun 2002, Wabah Canada pada tahun 2004 dan wabah di Mexico yang disebabkan H7N3 pada tahun 2012, kejadian meningkatnya virulensi adalah akibat dari insersi 30 nukleotida dari gen nukleoprotein, 24 nukleotida dari gen matriks dan 24 nukleotida dari RNA ribosomal yang berasal dari hospesnya yaitu ayam [54, 107, 154]. Kasus-kasus rekombinasi non-homolog lainnya telah terlihat pada studi eksperimental dimana sekuens nukleoprotein dan RNA ribosomal dari hospes diinsersikan pada situs pembelahan [60, 98]. Pada kelima contoh-contoh ini, kejadian insersi melibatkan beberapa asam amino dasar, tetapi jumlah mereka merupakan minoritas dibandingkan dengan keseluruhan jumlah insersi. Pada contoh-contoh ini meningkatnya jumlah celah pada loop situs pembelahan tampaknya merupakan faktor yang lebih penting yang dapat meningkatkan virulensi jika dibandingkan dengan hanya insersi asam amino dasar saja. Hampir seluruh virus H7 HPAI yang menyebabkan wabah tampaknya sebelumnya berubah menjadi sangat patogenik dikarenakan terjadinya kejadian unik pada situs pembelahan, yang menyebabkan sulitnya menentukan prediksi perubahan minimum virus H7 untuk menentukan perubahan menjadi HPAI jika hanya melalui prosedur sekuensing.
Variable-variable lain yang mempengaruhi patogenitas
HPAIV didefinisikan melalui test patotipe in vivo pada ayam, yang dapat diaplikasikan pada semua virus influenza dan atau dengan analisis sekuens dari situs pembelahan HA untuk H5 dan H7. Prediktor terbaik untuk HPAIV adalah jika virus terduga memiliki situs pembelahan yang sama dengan virus HPAIV yang telah dikenal. Pada situasi tersebut maka virus tersebut dapat dilaporkan kepada Organisasi Kesehatan Hewan Dunia (OIE) sebagai HPAIV. Meskipun begitu, sebuah wabah yang terjadi di Amerika Serikat (Texas) pada tahun 2004 adalah sebuah kasus yang jelas menunjukkan bahwa penotipe dan genotipe tidak menunjukkan kecocokan. Pada kasus ini isolat Texas/04 memiliki sekuens situs pembelahan HA yang sama dengan virus A/chicken/Scotland/59, dan sebelumnya telah dilaporkan sebagai HPAIV, akan tetapi virus tersebut merupakan Low Pathogenic (LP) berdasarkan uji penotipe standar pada ayam [79]. Meskipun kedua test tidak menunjukkan kecocokan dan virulensi tinggi tidak terlihat di lapangan, virus ini masih dikatakan virulen dan berakibat pada pemberlakuan sanksi perdagangan utama untuk eksportase unggas dalam periode terbatas. Contoh lain perbedaan fenotipe dan genotipe juga telah digambarkan sebelumnya [186] dan kasus yang serupa dengan virus H5 di Taiwan telah dilaporkan. Virus-virus ini memiliki empat asam amino dasar dimana terdapat beberapa virus yang bersifat patogenik berdasarkan testing IVPI dan ada beberapa virus yang tidak bersifat patogenik [74]. Sampai saat ini tidak ada skema prediksi molekular yang akurat untuk HPAIV.
Studi eksperimental dengan jelas menunjukkan bahwa umur dan rute inokulasi dan juga spesies dapat mempengaruhi virulensi virus A dalam situasi infeksi eksperimental. Efek dari umur telah terlihat pada ayam dan itik. Sebagai contoh saat anak ayam SPF umur 1 hari diuji tantang lewat intravena dengan virus LPAIV A/turkey/Oregon/1971, ditemukan kematian 7 dari 8 anak ayam. Saat virus yang sama diinokulasikan pada ayam umur 4 minggu dengan dosis dan rute yang sama, kematian hanya terlihat pada satu dari delapan ayam. Pada kasus ini, virus bereplikasi dengan jumlah titer yang tinggi pada ginjal yang berakibat gagal ginjal yang berlanjut pada kematian anak ayam umur satu hari. Virus yang sama diberikan melalui rute celah intra-choana (intranasal) menggunakan dosis yang sama menyebabkan kematian hanya satu dari delapan anak ayam umur satu hari [20]. Contoh ini mwnunjukkan bahwa kematian dapat sangat dipengaruhi oleh umur unggas dan rute inokulasi. Rute inokulasi intravena yang bukan merupakan rute inokulasi alami kemungkinan menempatkan virus dalam level sangat tinggi pada ginjal yang berakibat tingginya angka kematian. Tantangan beberapa virus LPAIV melalui rute intravena yang merupakan standar uji patotipe pada ayam dapat menyebabkan kematian sporadis, umumnya disebabkan replikasi pada ginjal yang berakibat gagal ginjal [134, 135, 163]. Sel ginjal primer ayam memungkinkan terjadinya replikasi virus LPAIV, mungkin karena sel-sel tersebut memproduksi enzim mirip tripsin yang dapat membelah protein hemaglutinin, dan hal ini memungkinkan timbulnya plak pada lajur cell ginjal embrio primer tanpa adanya penambahan tripsin [20].
Pada itik jika telah terlihat bahwa ada perbedaan gejala penyakit yang cukup signifikan berdasarkan umur hewan, dimana itik yang lebih muda lebih rentan terhadap infeksi berat. Sebagai contoh, beberapa virus H5N1 sal Asia menyebabkan kematian yang tinggi pada anak itik umur 2 minggu, tetapi virus yang sama hanya menyebabkan sedikit atau tidak ada kematian pada itik umur 4 minggu [104, 161]. Meningkatnya virulensi pada hewan muda umumnya terlihat, meskipun alasan perbedaan virulensi ini tidak jelas. Ketidakmatangan respons imun, baik bawaan ataupun adaptasi mungkin berkontribusi pada perbedaan ini. Sebagai contoh, respon interferon semakin meninggi mengiringi usia dari embrio. Kemungkinan respon interferon tertinggi juga terjadi setelah menetas [87].
Pada beberapa kasus, virulensi bisa saja lebih tinggi pada unggas yang lebih dewasa atau unggas yang sedang dalam masa produksi. Contoh yang umum terjadi adalah pada infeksi virus influenza mirip babi pada kalkun. Breeder kalkun yang sedang dalam masa produksi dapat mengalami penurunan produksi telur yang berat, tetapi untuk flok yang tidak dalam masa produksi unggas sering ditemukan menjadi positif secara serologis tanpa adanya gejala klinis [6, 34, 45, 155]. Peningkatan kematian juga telah sering terlihat pada petelur layer kalkun. Ditemukan adalanya peritonitis pada kuning telur setelah adanya infeksi LPAIV. Hal ini tidak terlihat pada unggas yang belum dewasa [200].
Antigenik drift dan antigenik shift
Virus IAV memiliki dia mekanisme utama dalam membuat keseragaman dalam populasi virus, yaitu tingkat mutasi yang tinggi dan kemampuan menyusun ulang (reassort) segmen gen [86, 174]. Kedua mekanisme ini memberikan kemungkinan bagi virus untuk berubah secara cepat dan beradaptasi, berkontribusi pada kemampuan infeksi pada spesies hospes yang baru. Kemampuan untuk bermutasi secara cepat dan beradaptasi bukan merupakan sesuatu yang unik dalam keluarga virus RNA, akan tetapi beberapa virus dapat mentoleransi perubahan sekuens dalam tingkat lebih tinggi pada beberapa gen virusnya. Virus IAV seperti yang telah dijelaskan sebelumnya, dapat berbeda sangat jauh dalam susunan sekuens asam aminonya, terutama pada glikoprotein permukaan, hemaglutinin dan neuraminidase [95]. Perbedaan sekuens asam amino ini berakibat adanya perbedaan antigenisitas, sedemikian rupa bahwa antibodi terhadap virus H1 IAV hanya akan menetralisir virus H1 saja dan tidak menetralisir virus dari subtipe IAV lainnya. Perbedaan antigenik ini berakibat munculnya pandemi yang telah membunuh lebih dari 40 juta orang [168]. Pandemi kedua dalam abad ini terjadi pada tahun 1957 dimana virus H1N1 digantikan oleh virus H2N2. Pandemi ketiga dimulai dari 1968 dimana virus H2N2 digantikan virus H3N2 [190]. Pandemi yang baru-baru ini muncul adalah influenza H1N1 yang muncul pada tahun 2009. Virus ini, meskipun merupakan satu subtipe dengan virus H1N1 yang bersirkulasi secara musiman, cukup berbeda secara antigenik untuk dapat beredar dalam populasi manusia dan pada akhirnya menggantikan virus H1N1 lama yang sudah muncul sebelumnya [138]. Asam muasal virus penyebab pandemi secara umum tidak dapat dimengerti secara jelas meskipun tampaknya kemunculannya berasal dari masuknya virus IAV yang benar-benar baru ke dalam populasi manusia atau dengan terjadinya penyusunan ulang (reassortment) antara strain virus manusia yang bersirkulasi dengan virus IAV pada hewan [190]. Virus H1N1 tahun 1918 tampaknya merupakan virus yang sama sekali baru, tetapi virus H2N2 dan H3N2 merupakan virus hasil reassortan yang telah berubah beberapa gen di dalanya, termasuk terutamanya gen HA [190]. Virus pH1N1 berkerabat dekat dengan virus flu babi (SIV) yang bersirkulasi di Amerika Utara, akan tetapi reassortment dengan virus lain yang tidak diketahui berkontribusi pada penambahan dua gen lain yang memungkinkan virus untuk bereplikasi dan ditularkan dengan baik apda manusia [138].
Contoh paling baik yang menunjukkan antigenik shift dalam sejarah kedokteran hewan adalah pada virus influenza kuda (EIV). Secara historis, kuda telah mengalami infeksi oleh virus IAV subtipe H7N7 yang tampaknya telah bersirkuasi pada populasi kuda dalam jangka waktu yang lama. Pada tahun 1963 sebuah subtipe baru muncul yaitu H3N8 yang menginfeksi kuda di seluruh dunia dan akhirnya menggantikan subtipe H7N7, dengan isolat terakhir virus H7N7 didapatkan pada tahun 1974 [24, 102]. Untuk babi di Amerika Serikat subtipe H1N1 sebelumnya merupakan strain influenza yang umum yang bersirkulasi dari tahun 1918 sampai akhir tahun 1990an. Akan tetapi sejak tahun 1998, virus H3N2 mulai diisolasi di Amerika Serikat. Virus-virus ini adalah reassortant tidak biasa memiliki gen H1N1 mirip SIV, gen virus mirip virus manusia, dan gen mirip AIV. Virus H1N1, H3N3, Ph1N1, dan bahkan virus hasil reasortant lainnya (H1N2 dan H3N1) saat ini semuanya bersirkulasi di Amerika Serikat [56, 57]. Karena terjadinya antigenik shift, vaksin untuk kuda dan babi perlu untuk diperbaharui dengan memasukkan virus-virus yang baru untuk dapat menciptakan perlindungan secara sempurna. Akan tetapi perusahaan-perusahaan pembuat vaksin saat ini tidak begitu rpoaktif dalam memperbaharui vaksin, salah satunya dikarenakan masalah perundang-undangan, dan banyak vaksin untuk kuda yang memasukkan virus H7N7 sebagai antigennya meskipun virus H7N7 sudah tidak bersirkulasi selama 35 tahun.
Untuk unggas, antigenik shift bukan merupakan masalah yang utama karena perunggasan secara komersial memiliki masa produksi yang pendek. Infeksi AIV sudah tidak umum terjadi maka unggas komersial tidak terekspose pada virus ini secara alami dan vaksinasi tidak dilaksanakan kecuali untuk virus HPAIV H5N1 di Cina, Mesir, Indonesia, Vietnam dan Bangladesh. Karena itulah sebagian besar unggas sangat rentan terhadap infeksi subtipe influenza yang saja. Detail lebih lanjut tentang vaksin influenza pada unggas dapat dibaca pada Bab 15.
Kesimpulan
Influenza tetap menjadi masalah kesehatan yang besar pada populasi unggas, babi dan kuda. Keprihatinan terbesar adalah infeksi HPAIV karena menimbulkan gejala berat dan berakibat negatif pada perdagangan unggas. Akan tetapi infeksi virus LPAIV juga menjadi perhatian karena dapat menimbulkan gejala dan juga menimbulkan kerugian produksi. Virus ini juga tersebar lebih luas daripada HPAIV, dan untuk virus-virus LPAIV H5 dan H7 akan terus ada ancaman terus menerus akan terjadinya mutasi menjadi HPAIV. Virus AIV sulit untuk dikontrol karena adanya reservoir hewan liar, kemampuan adaptasi virus, dan kurangnya sarana-sarana pengendalian penyakit yang baik. Masalah-masalah terkait flu babi terus menerus berkembang menjadi kompleks seiring dengan reassortment virus-virus AIV babi dan manusia menyebabkan kontrol lewat vaksinasi menjadi sulit. Virus Flu kuda juga terus menerus berubah secara antigenik, meskipun saat ini hanya ada dua galur utama virus yang bersirkulasi. Sarana vaksinasi yang ada sekarang ini tidak memberikan perlindungan jangka panjang, dan secara umum hanya memiliki kecocokan antigen yang rendah dengan virus yang bersirkulasi di lapangan karena vaksinnya tidak di perbaharui secara reguler. Usaha-Usaha untuk dapat memahami virus-virus ini dengan lebih baik dan melaksanakan penelitian-penelitian dalam rangka menemukan metode-metode baru pengendalian penyakit harus menjadi prioritas komunitas dokter hewan.
DISCLAIMER
Artikel terjemahan ini dimaksudkan bagi pembaca berbahasa Indonesia untuk dapat mempelajari artikel keilmuan terkait. Penerjemah tidak mendapatkan keuntungan apapun dari kegiatan penerjemahan ini.

