Oleh: Amy L. Vincent, Nicola Lewis and Richard Webby
Diterjemahkan oleh: Agna D. Lantria
* Penyebutan merek dagang maupun produk komersil dalam artikel ini dimaksudkan hanya untuk memberikan informasi secara spesifik dan tidak mengimplikasikan pemberian rekomendasi maupun endorsement oleh United States Department of Agriculture (USDA). USDA merupakan badan yang memberikan kesempatan yang sama, baik untuk penyedia maupun pemberi kerja.
DISCLAIMER
This article was translated to enable Bahasa Indonesia readers to better understand the topic explained inside the article in their native language. The translator made no profit in translating the article.
Pengenalan
Lebih banyak babi yang dikonsumsi di dunia dibandingkan dengan hewan penghasil daging lainnya di seluruh dunia, permintaan protein dari populasi manusia yang terus bertambah berarti produksi babi mau tidak mau akan terus berkembang [69]. Konsumsi daging diperkirakan bertambah dua kali lipat dalam 30 – 50 tahun kedepan, dan untuk memenuhi permintaan ini diperlukan babi dengan tubuh yang tidak berlemak dan bertumbuh dengan cepat yang tumbuh dengan baik dalam peternakan modern berkelanjutan dengan sistem kendali sesuai cuaca. Selain meluasnya produksi dan konsumsi babi di negara maju yang dimotori oleh meningkatnya permintaan daging secara global, seiring negara-negara menjadi lebih makmur permintaan dagingnya akan meningkat lebih tinggi lagi. Kenaikan populasi babi yang sangat dramatis mungkin akan membawa perubahan dalam evolusi virus influenza dengan segala kemungkinan yang tidak diketahui terhadap seluruh epidemiologi virus influenza A (IAV).
Informasi tentang virus AI dalam populasi babi secara umum memang relatif jarang ditemukan. Tetapi kebanyakan sekuens virus AI publik dalam GenBank [5] dari virus AI yang bersirkulasi dalam spesies hospes yang penting ini berasal dari negara maju. Virus AI tetapi menjadi penyebab dari salah satu penyakit pernapasan paling penting pada hewan dan manusia, akan tetapi terdapat kurangnya konsistensi antara surveilans virologi untuk melacak strain virus AI yang bersirkulasi dalam konteks kepadatan populasi babi (Gambar 18.1). Hal ini menghambat pemahaman kita tentang ekologi virus AI global secara keseluruhan, demikian juga resiko relatif penyebaran virus AI antar benua dan antar spesies dari babi. Wabah yang terus berulang dan penyebaran virus AI yang berbeda secara genetik dan antigeniknya secara cepat merepresentasikan tantangan besar untuk produksi unggas dan juga kesehatan masyarakat, tidak hanya dalam hal mengukur dan mengendalikan strain virus yang terus berevolusi pada babi, akan tetapi juga dalam mengkarakterisasikan potensi zoonosisnya [44].
Influenza babi pertama kali dikenali sebagai penyakit klinis yang bersamaan dengan pandemi flu spanyol tahun 1918, diikuti dengan isolasi dan karakterisasi virus AI babi di tahun 1930 [51, 93]. Virus HN1N klasik asal babi ini (cH1N1) bersirkulasi dalam banyak populasi babi di dunia, dalam beberapa kasus sampai mendekati 70 tahun dengan perubahan genetik yang sedikit [25]. Subtipe virus H1N1, H1N2, dan H3N2 saat ini endemik pada babi di seluruh dunia, memperlihatkan keragaman substansial tidak hanya dalam segmen gen hemaglutinin (HA) dan neuraminidase (NA) saja, tetapi juga dalam keenam segmen gen lainnya. Keragaman HA dan NA secara global disimpulkan dalam Gambar 18.2. Banyaknya subtipe dan keturunan genetik virus yang saling bersirkulasi bersama-sama memiliki leluhur yang berbeda-beda berkaitan dengan sejarah leluhur mereka yang dapat ditarik hingga hampir 100 tahun yang lalu.
Kebanyakan dari keragaman genetik virus AI pada populasi babi adalah akibat dari transmisi dua arah antara babi dan manusia [73, 76], diikuti dengan periode evolusi genetik dan antigeniknya, termasuk antigenic shift yang terjadi melalui reasortansi dalam tubuh babi. Kesempatan babi secara fungsional dan ekologi untuk berlaku sebagai ‘wadah’ percampuran dalam menghasilkan generasi virus AI yang baru telah berakibat munculnya arah evolusi virus-virus yang beradaptasi dengan babi secara dinamis, dengan adanya periode-periode perubahan genetik dan antigenik cepat yang diikuti oleh periode stabilitas [67]. Pada manusia, babi dan beberapa hospes avian, ekspresi reseptor asam sialik yang berfungsi sebagai tempat perlekatan virus serupa influenza di saluran pernafasan (𝛂2,6 dan 𝛂2,3) tampaknya memfasilitasi penularan dua arah ini [35], dan ketika varian masuk ke dalam hospes yang baru, faktor-faktor terkait populasi yang berbeda pada manusia dan babi menjalankan virus di sepanjang arah evolusi non-paralel.
Re-introduksi virus musiman H3N2 dan H1N1 dari manusia ke babi dalam titik waktu yang berbeda, di wilayah geografis berbeda, dengan adanya reasortansi dalam babi (termasuk di dalamnya gen internal), telah berkontribusi pada heterogenitas yang cukup signifikan terhadap keturunan genetik virus yang bersirkulasi pada babi secara global. Meskipun sering ada leluhur virus musiman asal manusia yang ada pada virus AI spesifik di wilayah tertentu pada babi, demikian juga penyebaran virus AI keturunan asal babi yang kadang-kadang terdokumentasikan antar benua, variasi genetik regional yang kompleks memungkinkan diskriminasi virus AI babi dari wilayah lain, dari virus influenza manusia yang saat ini beredar. Akan tetapi gambaran yang sebenarnya tentang virus AI yang bersirkulasi dalam populasi babi di dunia sulit untuk ditentukan, karena banyak wilayah kurang surveilansnya jika dibandingkan dengan level yang terlihat untuk virus influenza dalam populasi manusia. Memang virus AI dapat tidak terdeteksi sampai berdekade lamanya dalam populasi babi yang kurang jumlah sampelnya, dan berpotensi muncul dan menginfeksi spesies hospes lain.
Contoh paling dramatis dari keragaman genetis dan leluhur dari virus influenza yang seringkali leluhurnya tidak diketahui yang muncul seperti dicontohkan oleh virus pandemi H1N1 2009 (H1N1pdm09), suatu virus dengan segmen gen yang berasal dari dua virus AI babi dengan garis keturunan berbeda – H1N1 Eurasia dan virus tripel reasortan 𝜸-H1 Amerika Utara. Tetapi hubungan langsung dengan virus AI endemik babi tidak dapat juga dibuat, meskipun sudah dilakukan surveilans intensif lebih dari 5 tahun lamanya pada babi di seluruh dunia. Meskipun virus H1N1pdm09 muncul pada manusia di Meksiko dan menyebar dalam pandemi di seluruh dunia, diikuti dengan penularan dari manusia ke babi di beberapa wilayah [56], lokasi geografis dan spesies hospes dimana leluhur virus pre-pandemi berevolusi tetap menjadi misteri selama bertahun-tahun, sampai penelitian terbaru mengidentifikasi virus-virus pada babi di Meksiko yang secara genetis memiliki kekerabatan dekat dengan virus yang diduga sebagai prekursor pandemi [57a].
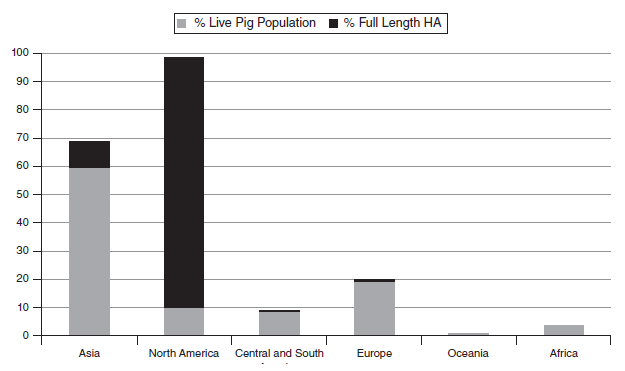
Gambar 18.1 Jumlah sequence gen hemagglutinin (HA) yang tersedia dalam database publik GenBank tidak konsisten dengan distribusi geografis dari populasi babi yang saat ini hidup. Meskipun Asia saat ini memiliki setidaknya 60% persen populasi babi di dunia, hanya 10% dari sekuens gen tersedia dari babi yang diisolasi sepanjang 2012-2013 yang berasal dari wilayah ini. Sebaliknya, Amerika utara memiliki sekitar 10 dari populasi babi dunia, namun melaporkan sekitar 90% sekuens HA AIV global yang berasal dari babi. Hanya ada sedikit sekuens yang tersedia dari babi di Amerika tengah dan selatan, Eropa dan Afrika. Wilayah Oseania hanya memiliki populasi babi yang rendah dan jumlah sekuens virus AIV dari babi yang rendah pula.
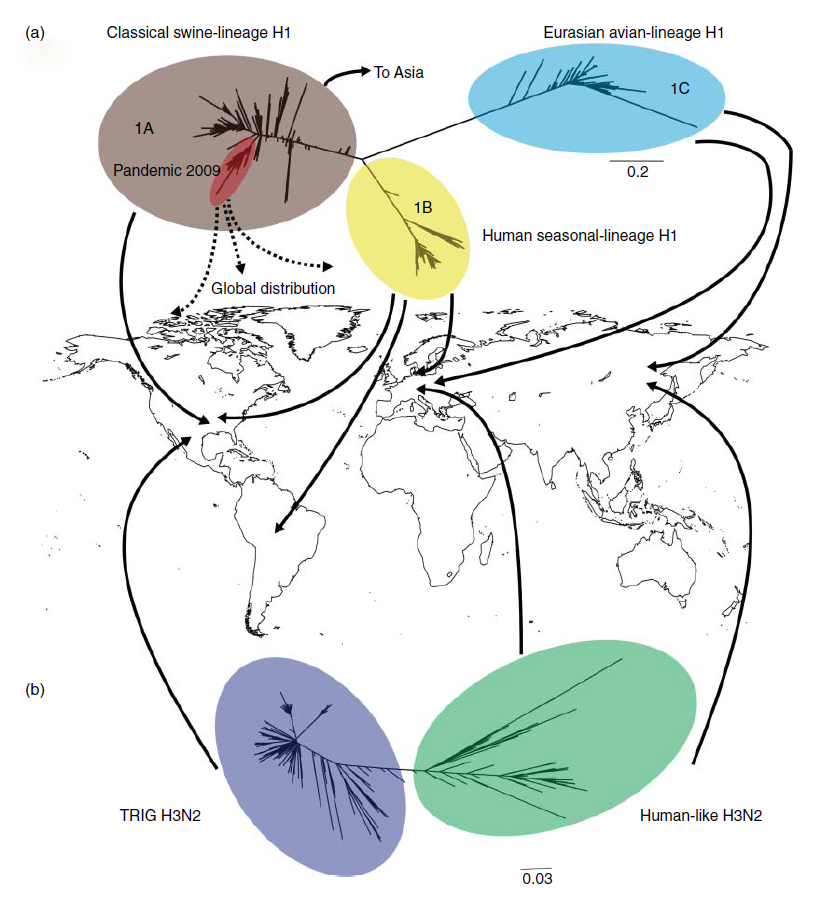
Gambar 18.2 Jalur keturunan H1 dan H3 utama dan distribusi geografisnya pada babi. (a) Hubungan filogenetik dari sekuens 1. (b) Sekuens H1 digambarkan dalam pohonnya masing-masing dan diberi kode warna berbeda sesuai jalur keturunan. Di Eropa, gen HA diturunkan dari virus H1N1 Eurasia Avian-like (ditunjukkan dengan warna biru), H3N2 human-like (warna hijau), atau H1N1 human-like (warna kuning). H1 klasik (warna coklat), H1 human-like (kuning) dan H3-TRIG human-like (ungu) bersama-sama berko-sirkulasi. Di Asia, jalur keturunan HA merefleksikan dinamika yang teramati di Amerika Utara dan Eropa, virus yang berko-sirkulasi diklasifikasikan sebagai jalur keturunan babi klasik, H3 human-like, atau H1 Eurasia avian-like. H1N1pdm09 muncul dari jalur keturunan H1 babi klasik, (warna merah), yang kemudian mengalami diseminasi secara global melalui penularan dari manusia ke babi. Digunakan dengan izin dari Vincent, A. L., K. . Lager, dan T.K. Anderson, 2-014. Pengenalan singkat tentang virus influenza A pada babi. Methods in Molecular Biology 116:243-258.
Distribusi geografis genotipe virus AI Babi
Amerika Utara
Amerika Utara meliputi tiga negara yang termasuk dalam 10 besar penghasil babi di dunia. Kebanyakan babi di Amerika dan Kanada dipelihara dalam kandang-kandang isolasi terkontrol dan dalam sistem yang terintegrasi, meskipun juga ditemukan pemeliharaan babi outdoor, peternakan belakang rumah dan/atau peternakan berskala produksi kecil, dan tipe peternakan yang terakhir ini lebih banyak ditemukan di Meksiko [78]. Operasional produksi babi meliputi farrow-to-finish, farrow-to-nursery, farrow-to-wean, wean-to-finish, dan finishing (fattening/penggemukan). Meskipun produksi babi dan/atau daging babi dapat ditemukan di sebagian besar wilayah Amerika Serikat, sejumlah besar babi yang telah disapih dipindahkan ke peternakan wean-to-finish di daerah Corn Belt di wilayah Midwest (Tengah Barat), karena peningkatan biaya pakan jika dibandingkan dengan biaya transportasi, dan ketersediaan pakan yang terbatas di wilayah lainnya. Terdapat trend yang signifikan menuju kepada konsolidasi industri ini sejak pertengahan tahun 1980-an, direfleksikan dari reduksi total jumlah operasi peternakan bersamaan dengan peningkatan tempat penyimpanan (inventory) oleh operasional peternakan yang lebih kecil. Meskipun terdapat 66 juta ekor babi yang dilaporkan pada tahun 2013, pada pertengahan tahun 2014-an populasi babi hanya sekitar 62,2 juta ekor babi saja di Amerika Serikat, kuat dugaan dikarenakan oleh wabah epidemi virus diare babi (porcine epidemic diarrhea virus) [107]. Amerika Serikat mengimpor sekitar 5 juta ekor babi per tahunnya antara tahun 2009-2013, terutama dari Kanada, dan mengekspor rata-rata 31.000 ekor babi per tahunnya dalam periode yang sama, dengan peringkat 3 besar pengimpor babi dalam 10 tahun terakhir adalah Meksiko, China Daratan, dan Rusia [105]. Di Kanada, populasi babi mencapai 12,7 juta pada tahun 2013 [101], dengan sekitar 5 juta babi hidup diekspor keluar terutama ke Amerika Serikat, dan hanya sedikit mengimpor balik [1]. Populasi babi hidup di Meksiko pada tahun 2013 kira-kira sebanyak 16,8 juta, dengan sekitar 15.000 ekor babi yang diimpor per tahunnya, dan hampir tidak ada laporan ekspor babi hidup [106]. Perdagangan babi hidup diantara negara-negara Amerika Utara dan juga dengan partner dagang jarak jauh mungkin telah memainkan peranan dominan dalam migrasi dan evolusi virus AI pada babi secara global [74].
Virus H1N1 klasik babi secara genetik stabil dalam populasi babi di Amerika Utara sampai tahun 1990-an [25, 51, 93]. Akan tetapi, di akhir tahun 1990-an, virus H3N2 triple reassortant baru diidentifikasi saat virus itu menyebar dengan cepat pada populasi babi. Virus baru ini mengandung segmen gen HA, NA, dan PB1 yang diturunkan dari virus H3N2 penyebab flu musiman pada manusia, segmen gen PB2, segmen PA dari virus AI avian, dan segmen gen NP, M, dan NS yang merupakan keturunan dari virus AI H1N1 babi klasik [123]. Kemudian, virus triple reasortan yang sukses ini mengalami reasortansi dengan virus H1N1 klasik, menciptakan keturunan baru virus H1N1 dan H1N2 [41, 42]. Kebanyakan kejadian reasortansi ini melibatkan hanya segmen gen H1 dan/atau N1 sja, melestarikan apa yang disebut sebagai konstelasi gen triple reassortant (TRIG) babi (segmen gen M, NP dan NS), avian (segmen PB2 dan PA), dan virus influenza asal manusia (PB1) [116]. Diantara kejadian reasortansi dramatis ini, keragaman genetik lebih jauh dibentuk oleh akumulasi mutasi yang berakibat perubahan pada protein permukaan virus (antigenic drift) [21].
Pada awal abad ke-21, virus H1N1 dan H1N2 dari babi mengandung baik gen HA, gen NA, atau kedua gen yang diturunkan dari virus influenza musiman asal manusia terdeteksi pada populasi babi di Amerika Utara dan Kanada [115]. Gen HA yang ditemukan secara genetik dan antigenik sangat berbeda dengan yang ditemukan pada virus H1 klasik yang diturunkan asal babi sebagai akibat dari antigenic drift yang dialami virus-virus H1N1 ini pada manusia. Pandemi H1N1 tambahan yang terjadi tahun 1977, diikuti dengan antigenic drift lebih lanjut yang terjadi pada manusia. Akan tetapi, gen internal TRIG dari virus H1N1 dan H1N2 babi yang diturunkan dari virus influenza musiman pada manusia serupa dengan yang ditemukan pada virus triple reassortant kontemporer. Ko-sirkulasi dari dua jalur keturunan virus H1 dengan variasi genetiknya telah menyebabkan perkembangan sebuah ‘klaster’ terminologi filogenetik di Amerika Utara. Virus dengan gen HA paling serupa dengan virus influenza H1 musiman pada manusia bersirkulasi pada awal tahun 2000-an membentuk klaster filogenetik 𝛅, dan yang lebih mirip dengan virus H1N1 klasik membentuk klaster 𝛂-, 𝛃- atau 𝜸- [115]. Lebih kompleks lagi, gen-gen HA yang membentuk klaster 𝛅 kemungkinan besar muncul dari setidaknya dua kali masuknya virus influenza musiman dari manusia, yang kemudian mengalami diversifikasi lebih lanjut di dalam populasi babi menjadi dua clade berbeda yang dinamakan 𝛅-1 dan 𝛅-2 [65], dengan 𝛅-1 menjadi clade dominan terdeteksi dalam waktu sekarang ini [4]. Keturunan gen HA ini ditemukan dalam gen N2 yang diturunkan dari virus AI musiman pada manusia baik dari 1998 atau 2002 vintage [75], atau dalam gen N1 sebagai keturunan virus babi klasik atau keturunan virus pandemi [4]. Sangat jelas bahwa Amerika Serikat dan Kanada berbagi keturunan virus Amerika Utara ini, meskipun dengan beberapa perbedaan regional dengan dominasi relatif, tetapi laporan dari Meksiko masih terbatas. Namun, berdasarkan data yang tersedia tampaknya virus pada babi-babi di Meksiko memperlihatkan beberapa tumpang tindih dengan setidaknya beberapa virus babi yang berasal dari keturunan Amerika Utara [63].
Keragaman virus AI pada babi di Amerika Utara semakin diperluas dengan terjadinya penularan dua arah (bidirectional) virus H1N1pdm09 asal babi, pada awalnya terdeteksi pada manusia di Meksiko dan kemudian diidentifikasi di seluruh dunia [30], dan kemudian mengalami introduksi kembali pada babi di Amerika Utara [34, 119]. Saat masuk ke tubuh babi, virus dari jalur keturunan H1N1pdm09 mengkontribusikan gen internalnya kepada subtipe endemik kedalam banyak kombinasi melalui reassortment [24, 48, 60], dengan gen matriks ditemukan pada mayoritas virus AI asal babi endemik di tahun 2012 [4]. Virus -virus hasil pada saat ini berada dalam periode evolusi genetik yang cepat, membawa ekspansi dari klaster IV virus H3N2 yang sebelumnya stabil menjadi beberapa sub-klaster yang bersirkulasi secara bersamaan, yang ditandai sebagai A – F [47, 57]. Masih menunggu untuk dilihat apakah semua tipe klaster H3 yang berkembang akan terus mengalami transmisi dan evolusi yang berkelanjutan, tetapi konsekuensi biologik dari keragaman genetik telah mulai muncul dengan dimanifestasikan oleh antigenic drift [29, 57]. Fenomena serupa tampaknya terjadi juga pada gen-gen HA dari virus-virus H1𝛅-1, akan tetapi bukti antigenic drift nya masih belum ada.
Amerika Selatan
Amerika Selatan melaporkan populasi babi sejumlah 62,3 juta ekor pada tahun 2013 [28]. Brazil adalah negara penghasil babi terbesar di Amerika Selatan, dan termasuk peringkat ke-3 di dunia dengan hitungan 38,6 ekor. Kebanyakan negara lainnya di Amerika Selatan memiliki populasi babi dengan kisaran 1 – 5 juta. Tipe sistem produksi sangat bervariasi di wilayah ini, dan lebih dari 50% populasi babo diperkirakan dipelihara secara tradisional dalam skala kecil di luar ruangan/outdoor atau dalam kandang di belakang rumah. Karena konsumsi daging sapi lebih besar daripada daging babi pada banyak negara di Amerika Selatan, produksi babi secara intensif cenderung terletak pada wilayah-wilayah dengan jumlah padang rumput untuk ternak sapi yang terbatas [38]. Meskipun begitu, populasi babi telah berkembang lebih dari 20% selama 10 tahun terakhir ini untuk memenuhi permintaan daging yang terus meningkat di wilayah ini dan karena ada potensi peningkatan pasar ekspor.
Hanya ada sedikit sekuens virus AI yang dapat diakses secara publik dari isolat babi yang tersedia dari negara-negara Amerika Latin. Argentina melaporkan keberadaan virus-virus dari subtipe H1N1 dan H3N2 dari keturunan virus manusia yang berbeda [12, 84]. Virus-virus Argentina dapat dibedakan dari subtipe-subtipe serupa yang ada di Amerika Utara dan mewakili kejadian penularan dari manusia ke babi yang terjadi secara independen. Di akhir tahun 2008, virus H3N2 yang secara penuh berasal dari manusia diisolasi dari babi yang menunjukkan gejala klinis penyakit pernafasan dan demam yang menciri pada infeksi influenza [12]. Di tahun 2009 dan 2010, Argentina melaporkan isolasi virus reasortan yang mengandung gen internal dari virus H1N1pdm09 dan gen permukaan (HA dan NA) dari virus AI H1 babi mirip-manusia (human-like) (mirip dengan virus Amerika Utara 𝛅2) [84]. Di tahun 2011, virus reasortan lain berhasil diisolasi, yang mengandung gen-gen permukaan yang berasal dari virus H3N2 secara penuh virus asal manusia yang pertama kali diisolasi di tahun 2008, dan gen internal yang berasal dari virus H1N1pdm09 [23]. Di Brazil, terdapat beberapa laporan infeksi virus AI pada babi sebelum tahun 2010. Baru-baru ini, bersamaan dengan virus H1N1pdm09 pada manusia, sejumlah wabah infeksi respirasi akut pada babi dari kelompok umur berbeda dilaporkan di Brazil, dan virus H1N1pdm09 diidentifikasi sebagai penyebabnya [89, 91]. Selain itu, virus AI H1N2 diidentifikasi dalam penelitian terbaru mengandung gen H1 dan N2 asal virus musiman pada manusia, dan gen internal (M, NP, PB1, PB2, dan PA) dari H1N1pdm09 [8].
Eropa
Uni Eropa (UE) melaporkan jumlah babi sebesar 147 juta ekor di tahun 2013, dengan kurang lebih 500.000 ekor dari ekspor, dengan Denmark menjadi eksportir terbesar. Negara eropa dengan populasi babi terbesar adalah Jerman dan Spanyol, dengan lebih kurang masing-masing sebesar 27 juta dan 28 juta ekor, diikuti oleh Perancis, Denmark, dan Belanda, dengan jumlah populasi masing-masing sekitar 12 – 13,5 juta ekor [28]. Produksi babi telah menurun di Eropa Barat secara umum, akan tetapi beberapa negara Uni Eropa telah mengalami sedikit perkembangan dalam tahun-tahun terakhir. Tipe dan ukuran sistem produksi di Eropa sangat bervariasi, dan Uni Eropa telah menuliskan secara mendetail arahan dalam beternak babi berdasarkan pertimbangan kesejahteraan hewan. Produksi babi berkisar dari peternakan berukuran kecil dengan jumlah 10 ekor babi atau kurang dari itu sampai peternakan breeding indoor dengan kapasitas 400 ekor babi, akan tetapi kebanyakan peternakan memiliki ukuran medium, dengan kapasitas 200 – 400 ekor babi dan menggunakan sistem farrow-to-finish [27].
Di Eropa, setidaknya terdapat tiga jalur keturunan virus utama yang sekarang ini beredar yang berbeda dengan jalur keturunan dan evolusi virus AI babi di Amerika Utara (direview oleh Brown [10] dan Vincent, dkk [113]. Meskipun terdapat sirkulasi ekstensif virus AI babi klasik H1N1 di pertengahan abad ke-20, virus-virus ini digantikan oleh jalur keturunan lain yang diturunkan langsung dari H1N1 asal unggas di akhir tahun 1970-an. Selain itu, leluhur paling umum dan paling baru dari virus H3N2 yang saat ini beredar pada populasi babi di Eropa adalah virus keturunan H3N2 yang merupakan virus pandemi pada manusia tahun 1968 (A/Hong Kong/I/68), dan karena itu berbeda dari virus H3N2 kontemporer di Amerika Utara, karena virus-virus H3N2 masuk dari manusia kepada babi setelah tiga dekade setelah pandemi tahun 1998 [110]. Virus-virus H3N2 ini bereasortansi dengan virus AI babi H1N1 mirip avian di pertengahan 1980-an, mendapat kaset gen internalnya [13]. Di Inggris, virus reasortan H1N2 baru dideskripsikan satu dekade kemudian, yang mengandung gen HA dari virus H1N1 asal manusia yang serupa dengan A/England-1980, dengan gen lainnya berasal dari virus AI H3N2 babi yang dominan bersirkulasi. Tiga jalur keturunan ini – H1N1 avian-like Eurasia, H3N2 human-like, dan H1N2 human-like – bersirkulasi bersama di Eropa. Meskipun jarang ditemukan, reasortan tambahan juga sudah terdeteksi, seperti misalnya virus-virus H1N1 dengan gen virus H1 avian-like dan virus-virus H1N2 dengan gen H1 human-like, juga gen-gen lainnya menyerupai prototipikal H1N2 Eropa [53].
Setelah masuknya virus H1N1pdm09 pada babi, keturunan endemiknya terus ber-ko-sirkulasi, memberikan kesempatan yang cukup untuk terjadinya antigenic shift and drift lebih jauh lagi. Dari tahun 2010 sampai 2013, program surveilans virologis yang ekstensif dilakukan oleh mitra-mitra lembaga European Surveillance Network for Influenza in Pigs (ESNIP3), sebuah konsorsium yang dibiayai oleh Uni Eropa [96]. Intensitas program surveilans sangat bervariasi antar negara yang terlibat, akan tetapi seringkali berkorelasi dengan level produksi daging babi (lihat diatas). Karena itulah, wilayah yang memiliki produksi intensif tidak terhindarkan lagi akan memiliki jumlah total kawanan babi yang diinvestigasi dalam jumlah yang lebih besar ketika program surveilans swasta/mandiri atau surveilans publik sudah ada dan berjalan, seperti misalnya di Jerman, Italia, Denmark, Perancis, dan Inggris. Kunjungan surveilans ke peternakan yang mengalami sindrom pernafasan akut dianjurkan oleh ESNIP3, dan karena itulah jumlah kunjungan meningkat selama program surveilans dilaksanakan. Karena hal ini pula terjadi kenaikan sebanyak hampir 45% jumlah kawanan babi yang diinvestigasi antara tahun k-1 dan tahun ke-3 dari program yang dilaksanakan ESNIP3. Kunjungan ke peternakan ini berakibat terdeteksinya virus AI dari 31% kasus (2759 kawanan positif dari total kawanan sebanyak 9025). Seperti halnya jumlah investigasi, frekuensi kasus positif juga sangat bervariasi tergantung pada negaranya, berkisar dari 3% sampai 67%. Akan tetapi, infeksi virus AI terkonfirmasi sepanjang tahun, tidak tergantung kepada musim.
Subtyping awal menunjukkan bahwa di kebanyakan negara, virus AI enzootik keturunan Eropa demikian juga H1N1pdm09 merupakan subtipe yang dominan [96]. Konsisten dengan hasil sebelumnya yang didapat selama berjalannya proyek ESNIP1 dan ESNIP2 yang dilaksanakan antara tahun 2000 dan 2008 [53, 54. 109], keturunan H1N1 babi avian-like (H1avN1) yang muncul di tahun 1979 adalah keturunan virus yang paling sering ditemukan di setiap negara, mewakili 53,6% subtipe yang diidentifikasi dalam periode tersebut. Sebaliknya, keturunan virus enzootik “reassortant H3N2 babi human-like” yang muncul di tahun 1984 hanya mencakup sebanyak 9,1% dari virus yang sudah diidentifikasi. Hasil ini konsisten dengan temuan ESNIP2, yang melaporkan bahwa virus ini tidak terdeteksi lagi di beberapa wilayah yang memiliki jumlah kepadatan populasi babi yang tinggi, sementara di sebagian wilayah Eropa lainnya masih lazim ditemukan. H3N2 bersirkulasi secara luas di banyak negara penghasil babi, seperti Belgia, Belanda, Jerman, Italia dan Spanyol, akan tetapi hampir seluruhnya tidak ada selama bertahun-tahun di Denmark, Inggris, dan Perancis. Keturunan virus enzootik H1N1 reassortant babi human-like (H1huN2) yang muncul di tahun 1992 mewakili keturunan genetik virus AI yang paling sering bersirkulasi di Eropa, dan telah teridentifikasi sebanyak 13% dari virus yang dikarakterisasi dalam studi ini. Selain itu, virus-virus reasortan antara tiga subtipe AI enzootik (misalnya rH1huN1 dan rH1avN2) terdeteksi sebanyak 7,2% dari seluruh kasus yang ada, di beberapa negara. Satu reasortan (rH1avN2) tampaknya telah menetap dalam populasi babi di Denmark.
Selama periode 2012-2013, virus-virus H1N1pdm09-like telah diidentifikasi di banyak negara, meliputi Jerman, Denmark, Inggris, Hungaria, Polandia, Italia, Perancis dan Finlandia. Sebaliknya, virus-virus ini tidak terdeteksi di negara lain yang selama ini mendeteksi kasus influenza dengan jumlah yang signifikan, seperti misalnya Belgia, Belanda dan Spanyol. Di Finlandia, virus H1N1pdm09 hanya terdeteksi secara sporadis beberapa bulan setelah berakhirnya pandemi pada manusia, akan tetapi pada kebanyakan negara virus ini telah diisolasi dengan frekuensi yang kian meningkat seiring waktu sejak tahun 2010. Virus ini juga terdeteksi di Israel, melalui program surveilans aktif, pada kumpulan babi tanpa adanya gejala klinis. Ko-sirkulasi H1N1pdm09 dengan virus AI enzootik H1N1, H1N2 dan H3N2 Eropa mengakibatkan berbagai kejadian reassortment, menyebabkan terdeteksinya virus baru hasil reasortansi yang sebagian besarnya telah saling berbagi gen HA dan/atau NA. Virus-virus ini meliputi sejumlah 6,5% dari virus-virus yang telah diidentifikasi, dan karena itulah berada hampir pada level yang sama dengan virus-virus hasil reasortan yang berada diantara strain enzootik lama. Jika ditotalkan, 16,8% dari virus-virus itu merupakan H1N1pdm09-like virus atau virus hasil reasortan yang telah mendapatkan satu atau lebih gen yang berasal dari H1N1pdm09 [96].
Asia
Asia mengandung sekitar 60% populasi babi di dunia, dengan jumlah 588 ekor [28]. Dalam kategori negara, China memiliki populasi babi terbesar di dunia, dengan estimasi 457 ekor di China Daratan dan 1,7 juta ekor dari ekspor yang dilaporkan tahun 2013, dominannya menuju Hong Kong [106]. Diikuti oleh Vietnam dan Rusia, yang melaporkan masing-masing sebanyak 26,2 juta dan 18,8 juta ekor di tahun 2013, sementara negara-negara lainnya seperti Korea Selatan, India, Thailand, dan Jepang masing-masing melaporkan sebanyak 8 – 10 juta ekor. Meskipun sistem produksi sangat bervariasi di Asia, lebih dari 50% populasi babi diperkirakan dipelihara dalam sistem produksi tradisional dari babi lahir sampai dewasa (farrow to finish) berskala kecil atau bahkan skala rumahan. Di China, industri babi telah menyaksikan perkembangan dramatis dalam produksi babi sejak tahun 1970. Peternakan breeder yang dioperasikan oleh pemerintah mungkin dapat memelihara 2000 ekor babi, biasanya dalam sistem peternakan farrow to finish di satu tempat saja (single-site) di wilayah yang padat populasi babinya [70]. Peternakan-peternakan ini seringkali mensuplai peternakan komersial dengan skala lebih kecil atau peternakan skala rumahan. Peternakan swasta atau semi-swasta komersial cenderung beroperasi menggunakan sistem modern yang mencakup 2 tempat, dan diperkirakan terdapat lebih dari 100 perusahaan produksi yang masing-masing memiliki 10.000 ekor babi. Rusia juga telah meningkatkan jumlah produksi babinya sejak tahun 2005, pada saat yang sama industrinya mengalami konsolidasi dan modernisasi peternakan [52].
Virus H1N1 babi klasik bersifat enzootik pada populasi babi di seluruh Asia (seperti China, Jepang, Thailand dan Vietnam). Virus AI pada babi di China adalah akibat dari masuknya virus antar benua yang terjadi melalui perpindahan babi asal Eropa dan Amerika Utara disertai masuknya virus musiman asal manusia. Setelah kejadian pemasukan ini, reassortment yang terjadi antara virus-virus keturunan klasik dan virus yang baru masuk mengakibatkan adanya subtipe antigenik yang serupa dengan ada di Eropa dan Amerika Utara, tetapi dengan kombinasi konstelasi gen internal unik dan genotipe HA dan NA dominan (direview oleh Vincent, dkk. [113]. Sebelum tahun 2009 di China, virus babi klasik bersirkulasi bersama sebagai virus-virus H1N2 yang membawa gen NA yang berasal dari virus asal manusia baru-baru ini, virus H3N2 asal manusia (A/hong Kong/168-like, A/Port Chalmers/I/73-like, dan A/Sydney/05/97-like), dan virus-virus H1N1 Eurasia avian-ike [31, 94]. Selain itu, keragaman genetik virus AI dalam kawanan babi di China meningkat karena perpindahan babi antar benua bersama dengan virusnya [112], mengakibatkan pemasukan, sirkulasi dan menetapnya virus-virus H3N2 asal Eropa di tahun 1999 dan H1N1 di tahun 2001, bersama virus TRIG asal Amerika Utara sekitar tahun 2002. Virus H1N1pdm09 juga masuk ke populasi babi di Asia dan bereasortansi dengan subtipe endemik di China [111], Jepang [68], dan Thailand [49]. Pola reasortansi yang terjadi pada H1N1pdm09 di China serupa dengan uang terjadi di Amerika Utara dimana keturunan NA dan HA yang sebelumnya endemik memiliki gen internal dari H1N1N1pdm09, sedangkan H1N1pdm09 dan gen permukaannya tampaknya tidak bersirkulasi dalam populasi babi berdasarkan studi surveilans jangka panjang yang ada [58]. Keturunan HA dominan di tahun 2012 yang dilaporkan melalui studi ini adalah H3 turunan asal manusia yang bersifat musiman dan H1 keturunan asal unggas Eurasia, dengan deteksi sporadis virus babi klasik dan H1N1pdm09.
Selain virus-virus mirip virus asal Amerika Utara dan Eropa yang bersirkulasi di Asia, ada beberapa jalur keturunan baru, seperti misalnya H1N1, H3N2, dan H1N2 ditemukan di Thailand sejak tahun 1980-an. Sebelum tahun 2009, virus-virus H1N1 di Thailand dimasukkan ke dalam klaster baik virus babi dari jalur keturunan HA klasik maupun HA Eurasia dengan pola reassortment genotipe yang spesifik [102]. Dari tahun 2000 sampai 2007, virus-virus H3N2 yang mengandung gen HA dan NA yang berasal dari jalur keturunan virus manusia baik dari turunan virus babi Eropa ataupun dari jalur keturunan virus influenza manusia musiman yang lebih terkini telah terdeteksi, dengan mengandung gen-gen internal yang berasal dari jalur keturunan virus babi Eurasia (PB1, PB2, PA, dan M) dan H1N1 klasis (NP dan NS). Selain itu, lebih kompleks lagi, virus H1N2 mengandung kombinasi dari gen-gen dari virus endemik H1N1 dan H3N2 mirip virus dari manusia [102]. Seperti wilayah lainnya, virus-virus H1N1, H1N2 dan H3N2 kontemporer Thailand baru-baru ini telah memasukkan berbagai segmen gen internal dari virus H1N1pdm09 [16, 87]. Begitu juga di Jepang, virus-virus dari jalur keturunan klasik H1 dan virus H3 musiman keturunan asal virus manusia telah bereasortansi dengan virus H1N1pdm09 [40], demikian pula dengan strain-strain endemik yang sebelumnya bersirkulasi pada babi di Korea Selatan [46, 80]. Selain itu, virus hasil reasortansi virus-virus dari jalur keturunan virus baru H1N2 [79] dan H3N1 [80] Eurasia yang berasal dari unggas baru-baru ini telah teridentifikasi di Korea Selatan.
Evolusi antigenik virus AI pada babi
Kebanyakan surveilans virus influenza babi berfokus pada segmen gen yang mengkode protein permukaan hemaglutinin (HA) karena HA adalah target utama respons imun dan merupakan komponen antigenik utama dalam vaksin influenza manusia dan babi. Penilaian hubungan antigenik antara HA yang berasal dari subtipe berbeda yang bersirkulasi dalam setiap hospes, dan pengetahuan tentang evolusi HA secara relatif dari waktu ke waktu adalah kunci untuk memahami pola global penularan virus, menilai risiko relatif kejadian masuknya virus baru dalam setiap arah, mengidentifikasi strategi pencegahan untuk masa depan, dan pada akhirnya untuk bersiap-siap menghadapi epidemi dan pandemi di masa depan. Teknik komputasi yang baru yang baru-baru ini telah dikembangkan untuk mengkuantifikasi dan memvisualisasi evolusi antigenik virus influenza dalam sejumlah hospes [99].
Karakterisasi potensi zoonosis virus influenza A babi, dan perkembangan metode pengendalian untuk meminimalisir resiko, keduanya memerlukan pemahaman dalam beberapa area kunci, meliputi karakterisasi antigenik terkini virus influenza yang bersirkulasi pada babi secara global, dampak imunitas populasi babi dan faktor produksi dalam evolusi varian virus, peranan terkait introduksi secara berulang virus-virus musiman asal manusia kembali ke dalam populasi babi mungkin memainkan peranan dalam memodifikasi resiko kemunculan kembali strain yang memiliki potensi pandemi. Kedekatan manusia dan babi dan kecenderungan virus untuk berpindah antara kedua spesies ini menciptakan lingkungan ekologi kompleks dan hubungan evolusi hospes-patogen dimana virus yang masuk ke dalam satu atau lebih hospes mungkin secara antigenik berevolusi dalam populasi baru ini dengan arah yang berbeda dengan yang ada pada populasi pendedah virus. Perbedaan evolusi virus antar hospes yang semacam itu mungkin saja memungkinkan munculnya varian antigenik dari waktu ke waktu sehingga populasi lain akan menjadi naif secara imunologi.
Sebelum pandemi 2009 yang disebabkan oleh virus yang berasal dari dua jalur keturunan virus AI babi, resiko zoonosis virus H1N1 dianggap lebih rendah daripada resiko introduksi subtipe yang benar-benar baru seperti H5N1 ke dalam populasi manusia. Dasar penilaian ini tidak diragukan lagi dibatasi oleh adanya penghentian surveilans virus pada babi dalam beberapa waktu, sehingga data yang ada terutama data antigenik menjadi sangat kurang untuk menilai evolusi relatif H1N1 dan subtipe lainnya dalam babi dan dalam hubungannya dengan imunitas populasi manusia. Surveilans influenza babi yang telah dilaksanakan utamanya melakukan assessment data genetik, dan sudah diketahui dengan baik bahwa masuknya virus influenza ke dalam populasi babi dengan kejadian yang terpisah dari banyak hospes mulai dari 1918 telah mengakibatkan heterogenitas genetik yang cukup jelas dalam keragaman virus antar wilayah geografis. Sebaliknya, di dalam populasi manusia mulai dekade-dekade yang lalu telah terstruktur dengan baik asesmen ciri-ciri antigenik dan genetik virus influenza A musiman yang mendekati sebenarnya (real time), dan juga telah terlaksana pembaharuan vaksin yang cepat dan berdasar keilmuan. Evolusi genetik virus influenza A pada manusia terus berlanjut, sementara evolusi antigeniknya bersifat selang-seling dan berkelompok/klaster, dengan satu klaster bersirkulasi dalam satu periode waktu, dan kemunculan klaster biasanya bertepatan dengan perlunya memperbaharui strain vaksin [99].
Evolusi genetik terus menerus semacam itu juga telah menciri pada virus-virus influenza yang bersirkulasi pada babi. Tetapi sejak pandemi 2009, riset telah dilakukan berdasarkan wilayah geografis tertentu untuk mengetahui karakteristik antigenik dari jalur keturunan virus yang sedang bersirkulasi pada waktu itu. Sebagai contoh, virus H3N2 mirip Wuhan-95 Y yang bersifat musiman pada manusia masuk kedalam populasi babi di Amerika dan diisolasi melalui investigasi diagnostik di tahun 1998 dan terus melalui upaya surveilans sampai sekarang ini. Di dalam populasi babi, virus-virus ini berevolusi menjadi klaster-klaster antigenik yang berbeda dan bersama-sama bersirkulasi secara bersamaan, yang tidak hanya bersifat antigenik yang berbeda satu sama lain tetapi juga secara spasial jauh dari arah evolusi manusia dan dari strain bibit yang awalnya berasal dari manusia. Dasar molekuler dari terjadinya antigenic drift pada babi memiliki kesamaan dengan influenza H3 pada manusia yang teramati dari tahun 1968 sampai 2003, akan tetapi dengan magnitudo drift yang terus menurun seiring waktu [50, 57]. Banyak varian antigenik asing yang tidak membentuk klaster juga telah teridentifikasi pada babi, yang mungkin mewakili keragaman klaster yang masih kurang banyak sampelnya atau kemunculan strain asing. Apapun faktor populasi hospes yang memunkinkan kemunculan keragaman antigenik semacam itu, terdapat perbedaan yang sangat mencolok antara varian antigen subtipe tunggal yang cenderung untuk bersirkulasi pada suatu waktu dalam populasi manusia, dan potensi keragaman antigenik yang saat ini ada pada babi [57].
Sejak tahun 1950 telah meningkat permintaan daging di seluruh dunia, yang setengahnya dapat teratasi oleh spesies ternak yang bertumbuh dengan cepat seperti misalnya babi yang memiliki tingkat konversi pakan yang efisien. Bersama dengan meningkatnya jumlah babi juga telah dilakukan intensifikasi, dimana hewan dipelihara di unit peternakan yang lebih sedikit jumlahnya namun berukuran besar. Pada awalnya intensifikasi ini berfokus di Amerika Utara dan Eropa, dimana jumlah ternak babi meningkat perlahan atau cenderung stabil. Di beberapa bagian dunia berkembang, sekitar 50% dari populasi babi masih dipelihara dalam sistem produksi tradisional berskala kecil yang didorong dari kebutuhan nafkah. Dampak dari intervensi pengendalian penyakit (misalnya vaksinasi), sistem produksi yang berbeda, kekebalan populasi sebelumnya, struktur umur dalam kawanan dan ras dalam penularan, maintenance dan evolusi antigenik virus influenza dalam populasi babi masih belum dimengerti dengan baik, akan tetapi sangat mungkin bahwa produksi babi modern menawarkan banyak titik kunci dimana intervensi terjadi yang mungkin dapat mempengaruhi dinamika epidemiologi virus influenza pada babi.
Hewan babi dalam ekologi virus AI dan interaksinya dengan spesies hospes lainnya
Meskipun unggas air liar adalah reservoir alami untuk virus AI, hospes mamalia termasuk babi mempertahankan jalur genetik virus AI yang beradaptasi dan dikenali berbeda dari jalur keturunan lainnya berdasarkan ciri genetik dan antigenik. Virus cH1N1 keturunan asal babi relatif stabil dalam level genetik dan antigeniknya di Amerika Serikat dalam waktu hampir 80 tahun, sampai terbentuknya segmen gen keturunan triple reassortant dari virus AI asal manusia, unggas dan babi di tahun 1998. Tetapi seperti yang telah dijelaskan sebelumnya di awal Bab ini, virus AI dari keturunan alternatif sudah muncul pada babi sebelum tahun 1998, dan tetap menetap sebagai keturunan yang dominan di berbagai wilayah dunia.
Kejadian banyaknya pemasukan virus dari keturunan asal unggas atau manusia secara independen secara sporadis terdeteksi pada babi selama periode ini, akan tetapi tidak selalu sukses dalam mempertahankan penularan jangka panjang, atau tetap terisolir secara geografis. Tetapi sejak kemunculan virus triple reassortant H3N2 pada tahun 1998 dan penularannya kepada populasi babi di Asia, keragaman genetik virus AI yang telah dikenali terus meningkat di seluruh dunia, sebagian besarnya akibat beberapa kali masuknya virus-virus H1 dan H3 musiman keturunan asal manusia yang diikuti dengan reassortment dengan virus AI babi yang telah endemik secara lokal [76]. Hal ini diikuti oleh kejadian penularan virus H1N1pdm09 dari manusia ke babi yang tidak dapat dihindari lagi setidaknya 50 kali secara global [73]. Sejak masuknya keturunan genetik baru, terus terjadi peningkatan reasortansi dan keragaman genetik virus [16, 40, 46, 48, 55, 58, 72, 73, 75, 79, 80, 87, 112]. Level surveilans pada babi meningkat di seluruh dunia setelah munculnya H1N1pdm09, dan terus mengungkap keberadaan virus AI di wilayah-wilayah yang sebelumnya tidak diketahui sebagai wilayah endemi virus AI dan/atau keberadaan keturunan gen baru yang sebelumnya tidak dikenali pada babi. Sangat penting untuk mempertahankan level surveilans ini selain juga terus memperluasnya ke wilayah-wilayah dan populasi yang masih belum direpresentasikan dengan baik.
Babi memainkan peranan penting dalam ekologi influenza [117], dan interaksi saling mempengaruhi antara manusia dan babi [20, 26, 71, 92, 95, 108], demikian juga antara babi dan kalkun domestik [7, 77, 85, 86, 121] mewakili niche tertentu yang sebagian besar ada tanpa gangguan dari virus AI yang berasal dari unggas air liar [76]. Tidak hanya hubungan antara babi dan manusia yang berpotensi memiliki resiko penularan antar spesies secara silang. Di Kanada, virus-virus H3N2 triple-reassortant yang memiliki kaset gen internal “triple-reassortant” mampu untuk menular antar spesies kepada kalkun dan burung puyuh. Dalam tubuh hewan unggas ini, virus-virus ini secara antigenik serupa dengan virus referensi 2005, tidak seperti virus H3N2 pada babi di Kanada, dimana beberapa varian antigenik H3 bersirkulasi bersama-sama [77]. Investigasi terhadap pola-pola evolusi divergen subtipe yang sama di dalam banyak spesies hospes memberikan kita sebuah kesempatan untuk memahami poros penggerak ekologi yang kompleks yang berada di balik dinamika virus influenza dalam tubuh hospes yang berbeda, dan pada akhirnya memberikan informasi yang memungkinkan kita menilai resiko masuknya virus-virus ini kedalam hospes lainnya, seperti misalnya manusia.
Penularan zoonotik virus AI asal babi yang telah beradaptasi pada manusia telah didokumentasikan selama bertahun-tahun sejak tahun 1918, dan secara umum mengakibatkan penyakit mirip influenza yang serupa dengan virus AI musiman pada manusia, dengan sedikit bukti adanya penularan berikutnya antar manusia ke manusia lainnya. Salah satu masalah mendasar terkait dengan keragaman genetik virus yang ditandai yang bersirkulasi dalam populasi babi adalah meningkatnya resiko relatif re-inkursi virus AI yang bersirkulasi pada babi kembali ke dalam populasi manusia, menggarisbawahi apa yang terjadi saat pandemi virus asal babi tahun 2009 [30], dan baru-baru ini dengan kemunculan virus varian H3N2 di Amerika Serikat tahun 2012 [26]. Antara tahun 2009 dan 2013, 348 orang di Amerika Serikat terinfeksi dengan varian H3N2 (H3N2v), dan kebanyakan isolat manusia ini mengandung 7 segmen gen dari virus H3N2 babi triple reassortant dan 1 segmen gen (gen M) dari keturunan virus H1N1pdm09 [15], yang ditunjukkan dengan lebih sederhana dalam Gambar 18.3. Dua kasus lain teridentifikasi tahun 2014, lagi-lagi adalah reasortan antara H3N2 dan H1N1pdm09 asal babi. Kebanyakan dari pasien-pasien ini memiliki sejarah paparan dan kontak dengan babi, kebanyakannya melalui pameran pertanian [26, 39]. Meskipun terdapat peningkatan deteksi virus H3N2 pada babi dengan penanda genetik yang sama dengan virus H3N2v pada manusia selama 2011-2012, hal ini tidak mungkin merupakan satu-satunya penjelasan terhadap peningkatan H3N2v pada manusia, karena juga terdapat peningkatan untuk virus H3N2 yang secara genetik berbeda [48, 75] dan dalam bagian virus H1 dalam kumpulan data surveilans USDA [4]. Demikian juga, terdapat banyak deteksi virus H3N2 dan H1N2 dari babi-babi yang dipamerkan pada pameran pertanian di Ohio selama tahun 2009-2011 [9]. Sebaliknya dengan H3N2v, hanya ada 21 kasus H1N1v atau H1N2v yang sudah terdokumentasikan sejak 2005.
Mungkin saja terdapat sifat-sifat virus spesifik yang memberikan kemampuan lebih pada virus H3N2v dalam menginfeksi manusia, dan virus H3N2v dapat menular melalui udara pada musang [82], standar model laboratorium untuk virus AI manusia. Akan tetapi, varian virus AI isolat manusia manusia dan babi juga menunjukkan kesuksesan menginfeksi dan menular antar musang dalam studi ini dan studi yang lain [80, 81, 122]. Faktor penentu lainnya adalah kemungkinan kurangnya kekebalan dalam populasi manusia terhadap subtipe virus AI yang telah berkembang secara independen pada babi jauh dari virus prekursor musiman manusia yang merupakan virus asalnya dan strain yang digunakan dalam vaksin virus musiman pada manusia [57]. Sejumlah proporsi remaja dan dewasa muda menunjukkan bahwa mereka memiliki antibodi reaksi silang terhadap H3N2v, akan tetapi anak-anak dan orang dewasa tidak punya antibodi protektifnya [14, 97]. Vaksin musiman manusia yang ada sekarang yang mengandung H3N2 tampaknya tidak melindungi terhadap H3N2v [33, 98]. Karena banyak dari kasus H3N2v adalah pada anak-anak yang berkontak dekat dan mengalami periode paparan jangka panjang pada saat penyelenggaraan pameran pertanian, seluruh faktor ini merujuk kepada sejumlah rangkaian kemungkinan yang secara kolektif mungkin dapat meningkatkan penularan virus H3N2v dalam kejadian spillover ini.
Faktor kompleks yang nampaknya terlibat dalam kasus-kasus yang berhubungan dengan pameran babi yang disebabkan varian virus AI tidak menghilangkan resiko virus H3N2v atau virus AI adaptasi babi lainnya untuk dapat menyebabkan epidemi maupun pandemi jika virus-virus tersebut mendapatkan kemampuan untuk menular dari manusia ke manusia, memberikan kesempatan untuk lebih jauh lagi bermutasi dan beradaptasi kembali ke dalam hospes manusia. Akan tetapi, penularan yang berkelanjutan dari manusia ke manusia masih belum teramati sampai saat ini. Virus H3N2v dengan segmen gen yang berasal dari virus H3N2 musiman asal manusia lebih dari dua dekade lalu kini telah beradaptasi pada babi, berlaku sebagai peringatan tentang pentingnya babi dan manusia dalam terciptanya generasi virus baru yang perlu diwaspadai untuk kedua spesies hospes ini. Pemahaman yang lebih baik tentang faktor-faktor yang diperlukan untuk adaptasi virus-virus di antara hospes dan antar hospes (manusia dan babi) sangat penting untuk memecahkan siklus tersebut, dan setiap upaya mitigasi haruslah dipertimbangkan dalam konteks sistem produksi hewan ternak, praktek-praktek antropogenik, dan ekologi virus [43].
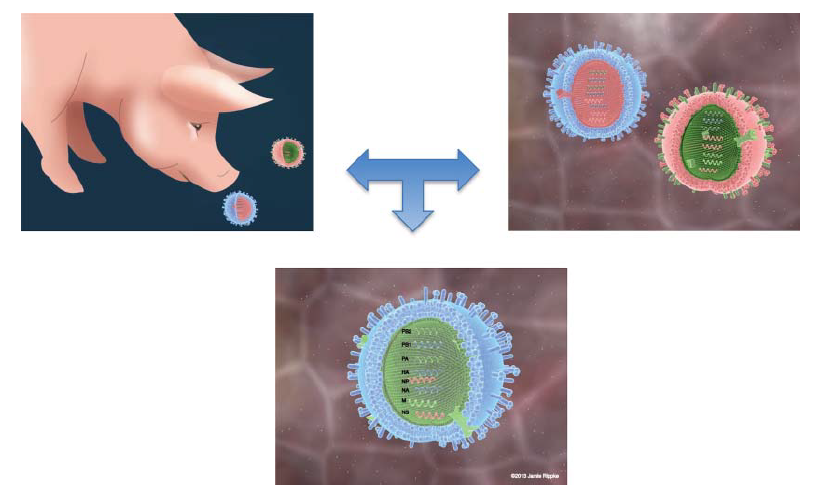
Gambar 1.3 Asumsi pembentukan varian H3N2 pada babi. Babi yang terinfeksi oleh dua strain virus AI – H3N2-TRIG (ditunjukkan dengan warna pink) – memungkinkan reasortansi konstelasi genom penuh secara spesifik dari masing-masing virus induk untuk membentuk progeni virion baru. Satu virus yang dihasilkan dari pola reasortansi potensial mengandung tujuh segmen gen yang diturunkan dari H3N2-TRIG dan satu segmen yang diturunkan dari H1N1pdm09 (gen M ditunjukkan dengan warna hijau terang). Virus H3N2 dan konstelasi genom yang diperlihatkan di sini telah ditemukan pada hampir 50 kasus pada manusia di Amerika Serikat sejak 2010, dan dinamakan varian H3N2 (H3N2v). Reasortansi antara virus babi endemik dan H1N1pdm09 telah sering terdeteksi pada populasi babi di seluruh dunia sejak 2009. Sumber: Amy Vincent dan Jamie Rippke.
Penentu virulensi dan cakupan hospes
Babi yang terinfeksi virus AI menunjukkan sejumlah spektrum penyakit klinis, berkisar dari tidak terdampak secara klinis sampai menunjukkan gejala demam dan gejala pernafasan parah. Kisaran gejala klinis ini mungkin disebabkan tingkat imunitas sebelumnya, karakteristik virus, dan banyak faktor kesehatan dan lingkungan lainnya. Gejala klinis influenza pada babi serupa dengan yang teramati pada manusia, dan termanifestasi sebagai penyakit saluran pernafasan akut, yang dicirikan dengan demam, inaktivitas, menurunnya jumlah pakan yang dimakan, kesulitan bernafas, batuk, bersin, konjungtivitis, dan leleran hidung [37, 90, 110]. Periode inkubasi penyakit adalah 1-3 hari, dengan kesembuhan cepat dimulai dari 4 – 7 hari setelah onset penyakit. Influenza babi dicirikan dengan morbiditas yang tinggi (mencapai 100%) dan mortalitas yang secara umum rendah (kurang dari 1%), dan dapat mengenai kawanan babi naif atau menyebar dengan lambat dalam populasi dengan status imunitas parsial atau campur. Sampai saat ini kita hanya memiliki pemahaman terbatas tentang kapan dan bagaimana babi bisa terinfeksi dalam tahapan produksi hewan ternak tertentu. Kunci untuk untuk menentukan dinamika penyakit dalam kawanan babi adalah pemahaman tentang varian virus yang bersirkulasi, stratifikasi umur dalam kawanan, profil imunologi babi dewasa dan anak babi, waktu infeksi dalam siklus produksi, dan peranan imun akibat vaksinasi atau akibat infeksi dalam menyebabkan beban influenza dalam kawanan. Virus-virus influenza juga umum ditemukan dalam kasus porcine respiratory disease complex (PRDC), bersamaan dengan patogen lainnya [103].
Influenza babi yang disebabkan oleh cH1N1 dalam sejarahnya dicirikan sebagai penyakit musiman, terutama pada babi sapih dengan imunitas maternal yang sedang memudar. Saat ini, penyakit klinis masih mencapai puncaknya pada saat-saat dalam setahun yang dihubungkan dengan fluktuasi dramatis dalam temperatur dan ventilasi yang meningkat [37], dan baru-baru ini telah menunjukkan puncak pertama pada bulan November dan Desember, dan puncak kedua di bulan Maret dan April [4]. Akan tetapi, penyakit dan diagnosis influenza dapat terjadi kapan saja dalam satu tahun, pada hampir seluruh kelompok umur, bahkan anak babi yang masih menyusu dari induk babi yang memiliki serum antibodi dengan titer tinggi terhadap influenza secara spesifik [3, 19]. Hal ini mungkin terjadi karena meningkatnya jumlah tipe klaster antigenik demikian juga variabilitas dalam transfer pasif dan populasi yang memiliki level dan spesifisitas imunitas yang saling bercampur. Meskipun antibodi maternal yang diturunkan secara pasif dapat mengurangi penyakit klinis, seringkali kurang efektif dalam mencegah replikasi dan penularan, terutama jika sudah ada antigenic drift dari virus-virus yang ada [2, 18, 104], menyebabkan babi-babi yang sudah disapih menginfeksi tempat persapihan dan/atau tempat finishing/penggemukan [3].
Faktor-faktor virologis yang mengendalikan infektivitas dan virulensi virus AI babi masih belum dijelaskan dengan baik jika dibandingkan dengan virus AI lainnya, sebagian besar disebabkan karena terbatasnya kapasitas studi eksperimental pada hospes alaminya. Kebanyakan data yang telah didapat untuk virus AI babi didapatkan dari model penelitian hewan kecil. Daripada bertujuan untuk menentukan persyaratan replikasi virus pada babi, studi-studi ini didesain untuk menentukan resiko zoonosis virus AI babi endemik. Studi oleh Ma dan koleganya telah menggarisbawahi pentingnya untuk sangat berhati-hati dalam mengekstrapolasikan data dari hewan kecil kepada babi, karena mereka telah menunjukkan bahwa dampak dari penanda patogenitas tikus yang telah dicirikan dengan baik, yaitu B2 627 terhadap keluaran penyakit pada mencit dan babi tidak berkorelasi dengan baik [66]. Tetapi, data yang telah terakumulasi dari studi semacam itu telah mengindikasikan bahwa virus AI babi berbagi banyak karakteristik serupa dengan virus AI manusia. Pada saat yang sama, meskipun virus influenza berpindah antara dua hospes ini, telah jelas bahwa terdapat perbedaan yang hampir tidak kentara diantara keduanya.
Sejauh ini, regulator paling ter karakterisasi dengan baik dalam seleksi hospes virus AI adalah interaksi HA dengan reseptor asam sialik pada sel hospes. Telah terdokumentasikan dengan baik bahwa babi memiliki reseptor untuk virus AI yang beradaptasi pada unggas dan mamalia, meskipun virus AI yang telah beradaptasi pada unggas yang dipasang pada babi menyebabkan berbagai varian dengan preferensi lebih besar terhadap reseptor virus mamalia [36]. Meskipun sudah jelas bahwa, jika diberikan dalam jumlah inokulum yang besar dan mencukupi, virus AI dapat bereplikasi pada babi [45], bukti epidemiologi secara jelas menunjukkan preferensi reseptor asam sialik 𝛂-2,6-linked diperlukan untuk memastikan kesuksesan terjadinya perlekatan, dan virus asal unggas secara umum kurang dapat beradaptasi pada babu [22, 59]. Meskipun baik virus AI yang telah beradaptasi pada manusia dan yang beradaptasi pada babi mengenali bentuk reseptor 𝛂-2,6-linked, sudah jelas bahwa kebanyakan infeksi zoonosis asal babi kemudian tidak diikuti dengan penularan berikutnya. Hal ini menunjukkan bahwa ciri molekuler optimal yang diperlukan oleh virus influenza untuk dapat menginfeksi babi tidak selalu sama dengan yang diperlukan untuk menginfeksi manusia. Dalam salah satu dari sedikit studi yang telah mempelajari tentang dampak penanda molekuler terhadap infektivitas virus AI terhadap babi, Busch dan koleganya meneliti kemampuan virus-virus H3N2 asal babi dan asal manusia untuk menginfeksi dan bereplikasi dalam sel epitel respirasi primer babi [11]. Virus asal babi lebih infeksius pada babi, dan juga pada sel epitel primer babi, jika dibandingkan dengan virus asal manusia. Perbedaan genetik yang bertanggungjawab terhadap hal ini ditemukan di dalam gen HA. Dengan menggunakan sejumlah virus mutan, kelompok peneliti ini telah berhasil menemukan fenotip diferensial yaitu dalam residu tiga asam amino. Meskipun kedua virus ditemukan berikatan dengan reseptor asam sialik 𝛂-2,6-linked, residu HA yang dimaksud tampaknya lebih mempengaruhi interaksi samar antara virus dan reseptor, meliputi preferensi aviditas dan konformasional [6]. Peneliti menyimpulkan bahwa interaksi diferensial terhadap asam sialik inilah yang paling dekat berkorelasi dengan infektivitas sel epitel babi, dan dengan itu dapat mempengaruhi kemampuan virus AI manusia untuk menginfeksi babi, dan kemampuan virus AI babi untuk menginfeksi manusia.
Tema-tema konsisten juga muncul saat virus-virus H1N1pdm09 yang diisolasi dari manusia diinokulasikan secara eksperimental ke dalam tubuh babi. Dalam satu penelitian, teramati dua perbedaan dalam kemampuan dua virus untuk bereplikasi dan menular kepada babi. Dengan menggunakan virus rekombinan, substitusi S186P pada HA ditemukan bertanggung jawab dalam meningkatkan efisiensi pertumbuhan. Penulis menyimpulkan bahwa “efisiensi ini muncul sangat mungkin melalui keuntungan dalam perlekatan permukaan sel dibandingkan dengan efisiensi replikasi” [64]. Polimorfisme 186P terlihat lebih sering pada virus-virus H1N1pdm09 yang diisolasi dari babi, menunjukkan bahwa polimorfisme ini telah terpilih dalam hospes ini melalui infeksi dan penularan alami.
Data lebih lanjut menunjukkan bahwa perubahan yang tidak kentara meregulasi fenotipe virus AI yang secara diferensial mampu untuk menginfeksi dan menulari babi dan manusia datang dari penelitian yang mempelajari aktivitas saling menyeimbangkan dari HA dan NA. Meskipun terdapat variasi dalam aviditas individu HA untuk reseptor seluler nya dalam virus-virus H1N1 yang berhubungan dekat dengan manusia, terdapat aktivitas NA yang seimbang, karena itulah HA yang lebih avid telah selalu berpasangan dengan NA yang lebih aktif [120]. Serupa dengan itu, virus manusia dengan HA beraviditas rendah selalu berpasangan dengan NA yang kurang aktif. Kebalikan dari hasil yang didapat dari virus yang diisolasi dari manusia, tidak dapat keseimbangan aktivitas HA dan NA pada virus H1N1 babi yang telah diuji. Masih belum jelas mengapa ketidakseragaman ini terjadi, akan tetapi data-data ini menunjukkan bahwa faktor yang mengatur perlekatan virus dan pelepasannya berbeda pada manusia dan babi. Menariknya, telah diberikan saran bahwa mucin pada babi telah berkurang kemampuannya untuk menghalangi perlekatan virus AI ke sel epitel dibandingkan dengan mucin manusia [17]. Seperti yang telah diperkirakan bahwa peranan NA adalah mengurangi hambatan virus yang tercapai melalui mucin, data-data ini konsisten dengan berkurangnya keperluan virus untuk mengoptimalisasi aktivitas HA dan NA pada babi.
Perubahan molekuler dalam protein virus lainnya telah dihubungkan juga dengan meningkatnya infektivitas virus pada babi, meskipun mekanisme di baliknya secara umum masih belum didefinisikan dengan baik. Sebagai contoh, Londt dan koleganya mencoba untuk mengadaptasikan virus AI H5N1 asal avian/unggas kedalam sel babi di bawah kondisi tekanan imunitas bawaan dan perolehan. Dalam adaptasi itu ditemukan mutasi pada protein PB2, PB1, HA, NP dan M, dan pasase virus berhasil mencapai titer tertinggi dalam sel kultur dan jaringan explans babi dalam media [62]. Meskipun perubahan molekuler penting tidak diidentifikasi secara definitif, penelitian semacam ini berhasil mengidentifikasi protein virus mana saja yang harus berubah agar virus asal unggas menjadi lebih efisien dalam menginfeksi babi. Perlu untuk digaris bawahi bahwa dalam satu studi yang sama, para penulisnya tidak dapat mengadaptasikan virus H7, hasil ini menunjukkan bahwa virus AI dengan subtipe berbeda mungkin memerlukan mutasi adaptif yang berbeda pula, dan mungkin beberapa diantaranya dapat menginfeksi babi secara alami. Menggunakan strategi yang sama, Wei dan koleganya mem pasase virus rekombinan yang didesain untuk menyerupai konstelasi genotip dari virus pandemi 2009 pada babi. Setelah pasase, dipilih substitusi dalam populasi virus untuk HA (dengan varian berbeda yang muncul pada saluran pernafasan atas dan bawah) dan PB1, PA, NA, NS1, dan NEP [118]. Konsekuensi fungsional dari perubahan-perubahan ini meliputi peningkatan aktivitas polimerase dan juga meningkatkan replikasi dan transmisi dalam berbagai model hewan coba, termasuk babi dan musang. Menyokong peranan pentingnya gen-gen non-HA dan non-NA dalam adaptasi virus AI pada babi, dilakukan penelitian yang menciptakan reasortan yang dihasilkan dalam laboratorium yang kemudian diuji kemampuannya dalam menginfeksi, menimbulkan penyakit, dan menularkannya pada babi. Dalam salah satu penelitian itu, virus reasortan dimana HA dan NA nya diturunkan dari virus H9N2 dan gen segmen lainnya diturunkan dari virus H1N1pdm09 mampu tumbuh dalam titer tinggi in vitro dan in vivo dibandingkan dengan virus induknya (H9N2) [32]. Data-data ini konsisten dengan temuan pada penelitian serupa yang telah dilakukan sebelumnya [88].
Seperti yang umum ditemukan (tipikal) pada endemi virus AI di populasi hospes lainnya, terdapat variasi yang cukup besar dalam patogenisitas isolat individual pada babi, bahkan dalam satu jalur keturunan monofiletik tunggal [misalnya 90, 100]. Akan tetapi, beberapa penelitian telah memperluas observasinya sampai tingkat determinan molekulernya. Pendekatan yang lebih umum dilakukan adalah memeriksa dampak penanda patogenitas yang telah diidentifikasi pada virus AI lainnya, paling sering berasal dari unggas, dalam virus AI. Contoh dari pendekatan ini adalah penelitian yang telah menyelidiki dampak restorasi open reading frame PB1-F2 dalam virus H1N1pdm09 (virus ini memiliki trunkasi yang terjadi secara alami) [83]. PB1-F2 open reading frame tidak terdapat pada banyak virus AI babi. Dalam penelitian ini, restorasi open reading frame memiliki dampak yang minimal namun terukur terhadap replikasi virus dan patogenitas virus H1N1pdm09 pada babi, yaitu terjadinya peningkatan kedua faktor tersebut. Lui dan koleganya juga telah meneliti efek polimorfisme pada posisi yang telah dikenali dengan baik dalam PN2 pada virus endemik asal babi [62], dan menemukan dampak terukur dalam pertumbuhan virus dan munculnya penyakit. Pendekatan semacam itu penting dan telah memberikan gambaran tentang patogenitas virus AU akan tetapi mereka tentunya tidak mengungkapkan penanda spesifik pada babi. Meskipun pandemi 2009 telah meningkatkan ketertarikan dalam mengetahui infektivitas virus AI dan penanda patogenitasnya, pengetahuan tentang hal ini masih sangat awal. Tanpa adanya pemahaman dasar tentang residu spesifik dan bagaimana mereka mempengaruhi fenotipe virus, kita tak akan mampu bahkan untuk berfikir memprediksi dampak diversifikasi genetik virus AI yang besar yang saat ini terjadi pada populasi babi global. Situasi ini harus berubah.
Kesimpulan
Surveilans, investigasi epidemiologi, dan ciri genetik virus AI yang berhubungan dengan wabah penyakit respirasi pada babi sangat penting untuk melakukan pengawasan evolusi virus-virus ini dalam populasi babi. Selain itu, karakterisasi antigenik diperlukan untuk mengerti secara penuh relevansi perubahan genetik sebagai dasar seleksi antigen untuk vaksin, dan efektivitas vaksin harus dievaluasi dalam konteks reaktifitas serologis silang saat varian baru muncul. Peningkatan surveilans untuk virus AI dan varian baru H1N1pdm09 dan virus AI endemik pada babi dalam populasi babi dan manusia sangat penting untuk memahami dinamika ekologi virus AI dalam populasi hospes yang rentan. Siklus penularan dari manusia ke babi, diikuti dengan evolusi virus pada babi, dan kemudian masuk kembali ke dalam populasi manusia telah menjadi tetap pada virus AI manusia dan babi yang ada pada saat sekarang ini [72, 75, 76]. Penularan virus AI antar spesies dua arah dan evolusi virus-virus ini yang terus terjadi pada babi dan manusia tidak pernah diprediksi sebelumnya dalam sejarah virus AI.
Meskipun proyek-proyek surveilans regional masih terus berjalan dan didukung oleh berbagai jaringan penyandang dana sementara (misalnya United States DHHS-NIH NIAID Centers for Excellence for Influenza Research and Surveillance (CEIRS), USDA IAV-Swine Surveillance System, ESNIP3, dan Thailand-Japan Zoonotic Disease Collaboration Center Swine Surveillance Project, diantara banyak yang lainnya),kebanyakan surveilans lainnya pada babi telah dilaksanakan dalam skala yang lebih kecil oleh investigator penyakit hewan individual. Untuk mengatasi kekurangan surveilans AI pada babi yang terintegrasi baik regional dan global, OIE dan FAO membentuk OFFLU (Jaringan ahli influenza hewan OIE-FAO) di tahun 2005 untuk mengatasi masalah avian influenza dan memperluas jaringannya di tahun 2009 dengan memasukkan keahlian terkait virus AI yang bersirkulasi pada babi, kuda dan hospes hewan lainnya. Misi OFFLU dicapai dengan kolaborasi dan pertukaran informasi, materi biologis, dan sumber daya antara seluruh sektor influenza. OFFLU Swine Influenza Virus Group terus proaktif dalam berbagi dan menganalisa virus-virus influenza dan sekuensnya yang didapat melalui aktivitas surveilans pada populasi babi di seluruh dunia [113]. Aktivitas yang baru-baru ini dilaksanakan meliputi penggunaan virus-virus musiman asal manusia dan virus-virus influenza babi yang saat ini bersirkulasi secara endemik untuk mengukur hubungan antigenik antara virus-virus yang bertanggungjawab terhadap munculnya kasus influenza pada babi dan manusia [70a]. Pengukuran hubungan antara berbagai varian virus influenza yang bersirkulasi pada babi dan manusia, dan pemahaman evolusi relatifnya dalam hospes-hospes ini seiring waktu, merupakan kunci baik dalam peningkatan vaksin untuk babi dan manusia, dan dalam memahami resiko relatif kejadian penularan antar spesies di masa depan. OFFLU Swine Influenza Virus Group juga bekerja untuk membuat sistem penamaan klaster HA secara umum untuk digunakan oleh sektor kesehatan hewan dan manusia dalam penyebutan virus influenza babi dalam skala global. Sistem ini akan memungkinkan evaluasi virus-virus di seluruh dunia dengan merujuk pada satu kriteria terstandar akan memungkinkan hubungan genetiknya untuk dapat dimengerti dalam suatu konteks yang sama. Sistem semacam itu juga penting dalam menargetkan kelompok-kelompok virus untuk dipelajari, baik ciri-ciri antigenik dan untuk mengembangkan vaksin yang efektif.
Meskipun virus AI pada babi telah didokumentasikan dan dipelajari selama hampir satu abad lamanya, banyak pertanyaan yang masih belum terjawab. Selain itu pula, program yang terintegrasi secara efektif dan sistematik dalam melaksanakan monitoring dan pencegahan infeksi AI pada babi masih sangat kurang baik di level regional, nasional maupun global. Perkembangan yang muncul akhir-akhir ini dalam program surveilans regional, metodologi sekuensing, penelitian dan kolaborasi melalui jaringan telah menetapkan sebuah kerangka kerja dimana akan terus dilakukan upaya pengendalian terus menerus untuk mengendalikan virus ini pada babi melalui praktek-praktek pemeliharaan yang baik dan atau memperbaiki produksi vaksin. Peningkatan dalam kesediaan sekuens dan penggunaan alat analisis yang lebih baru telah menunjukkan bahwa adanya kecenderungan virus-virus musiman pada manusia untuk mengalami tumpahan (spill over) ke populasi babi, demikian juga adanya potensi zoonosis virus-virus asal babi. Virus H1N1pdm09 menggarisbawahi pentingnya monitoring evolusi virus AI pada babi secara global dan melakukan penilaian resiko yang dimiliki virus-virus ini melalui model influenza manusia. Dengan begitu seluruh upaya berkelanjutan yang saat ini tengah dilakukan dalam sektor peternakan babi dan keterlibatan sektor influenza pada manusia akan menghasilkan manfaat ganda dalam meningkatkan kesehatan hewan dan kesehatan manusia.
DISCLAIMER
Artikel terjemahan ini dimaksudkan bagi pembaca berbahasa Indonesia untuk dapat mempelajari artikel keilmuan terkait. Penerjemah tidak mendapatkan keuntungan apapun dari kegiatan penerjemahan ini.

